- Accueil
- Volume 19 (2015)
- Numéro 2
- Biologie, diversité et outils pour l’analyse de la diversité génétique de l’oignon, Allium cepa L. (synthèse bibliographique)
Visualisation(s): 0 (0 ULiège)
Téléchargement(s): 0 (0 ULiège)
Biologie, diversité et outils pour l’analyse de la diversité génétique de l’oignon, Allium cepa L. (synthèse bibliographique)

Notes de la rédaction
Reçu le 12 juin 2014, accepté le 24 février 2015
Résumé
Description du sujet. Cette étude synthétise les principaux résultats sur la biologie de l’oignon, les ressources phytogénétiques, la position taxonomique ainsi que les marqueurs morphologiques, biochimiques et moléculaires pour l’analyse de la diversité génétique de l’oignon, avec une attention particulière pour l’Afrique. Plante monocotylédone, allogame, entomophile, avec un cycle cultural annuel pour la production des bulbes, bisannuel pour celle des graines, l’oignon est l’un des légumes les plus importants au monde en raison de son utilisation en alimentation et en médecine.
Littérature. Afin de caractériser la diversité génétique de l’oignon, 28 marqueurs morphologiques ont été définis sur base de l’observation des graines, des feuilles, des fleurs et des bulbes. Des marqueurs biochimiques et moléculaires ont aussi été développés, notamment pour analyser les variations génétiques entre et à l’intérieur des variétés. Des études entre les variétés de l’oignon d’Afrique de l’Ouest ont montré que seules six enzymes sont polymorphes. Toutefois, 24 enzymes ont été utilisées pour analyser la diversité génétique entre l’espèce Allium cepa et les autres espèces du genre Allium. Ce nombre limité de marqueurs biochimiques polymorphes rend difficile l’analyse de la diversité génétique chez l’oignon. Les marqueurs moléculaires directement issus du polymorphisme existant au niveau de l'ADN, comme RAPD, RFLP, AFLP, SSR, sont très utiles pour différencier les différentes variétés de l’oignon, pour distinguer l’espèce des autres espèces cultivées et spontanées du genre Allium et pour analyser les niveaux d’introgression entre l’oignon et d’autres espèces du genre.
Conclusions. L’analyse de la diversité génétique montre une importante variabilité entre et à l’intérieur des écotypes d’oignon en Afrique. Il est donc utile de combiner des stratégies de conservation in situ et ex situ, et l’exploitation de ces ressources génétiques pour améliorer leurs productions et leurs rusticités.
Abstract
Biology, diversity and tools for diversity analysis of the onion (Allium cepa L.). A review
Description of the subject. This study explores important information on biology, genetic resources, taxonomy, as well as morphological, biochemical and molecular markers in order to provide a better understanding of the genetic diversity of the onion (Allium cepa L.). The study has a particular focus on the plant as grown in Africa. The onion is a monocotyledonous, allogamous, and entomophilous plant, with a one-year production cycle for bulb production, and a two-year production cycle for seeds. The onion is one of the most significant vegetables in the world because of its use as both a food and a medicine.
Literature. Twenty-eight morphological markers, linked to seed, leaf, flower and bulb traits, were identified as the most discriminant phenotypical criteria. Biochemical and molecular markers were also developed to characterize genetic variations between and within onion varieties. Previous studies examining West Africa onion varieties showed that only six enzyme systems are polymorphic. However, only twenty-four isozymes have been used to compare the onion to other Allium species. This low number of polymorphic biochemical markers makes it more difficult to determine the genetic diversity of onions. On the other hand, molecular markers at DNA level, such as RAPD, RFLP, AFLP, SSR, are very useful to analyze diversity at varietal and species level, using cultivated and spontaneous forms, and to analyze the level of introgression between the onion and the other species of the genus.
Conclusions. Genetic diversity analysis showed an important variability between and within Africa onion landraces. It would be useful to combine in situ and ex situ conservation, using these genetic resources to improve the production and the appropriate use of African onion cultivars.
Table des matières
1. Introduction
1Les espèces alimentaires, oignon (Allium cepa L.), oignon multipliant (Allium fistulosum L.), ail (Allium sativum L.) et poireau (Allium ampeloprasum L.) sont des monocotylédones herbacées appartenant à la famille des Alliacées (Fritsch et al., 2002). La domestication de l’oignon s’est accompagnée au cours du temps d’une sélection de cultivars ayant un développement important du bulbe au cours de la première année de culture. Connu des Égyptiens, des Romains et des Grecs, cette espèce fut d’abord exploitée comme plante médicinale avant de devenir un condiment ou légume (De Lannoy, 2001). Sur la liste des légumes les plus cultivés au monde, les oignons sont classés deuxième, précédés par les tomates. Les oignons sont produits partout dans le monde, à différentes latitudes entre 10°S et 65°N (Foury et al., 1992). En 2012, la production mondiale de l’oignon fut de l’ordre de 82,85·106 t par an, avec 56,73·106 t par an pour l’Asie. Les principaux pays producteurs sont la République Populaire de Chine avec 22 600 000 t par an, l’Inde avec 16 308 990 t par an et les États-Unis avec 3 277 460 t par an. Sur le continent africain, les principaux pays producteurs d’oignon sont l’Égypte avec 2 024 881 t par an, l’Algérie avec 1 183 268 t par an, le Maroc avec 855 764 t par an et le Niger avec 382 000 t par an (FAOSTAT, 2013).
2Selon Currah (2002), les différentes variétés de l’oignon sont très sensibles à la longueur du jour. Elles sont généralement regroupées en trois categories : les variétés de jours courts, de jours moyens et de jours longs avec des besoins respectivement de 8 à 12 h, de 13 à 14 h et de 14 à 16 h de lumière par jour pour la bulbification. Dans les conditions de culture d’Afrique tropicale, les variétés de jours courts sont les mieux adaptées (De Lannoy, 2001). De nos jours, on peut trouver en Afrique des oignons avec des couleurs, gouts et formes de bulbes variés. Ils sont utilisés comme condiment dans plusieurs types de plats soit cuits en ragout, soit frits avant d’être consommés (Foury et al., 1992). Cependant, ils sont aussi mangés crus, particulièrement les oignons sucrés qui ont un gout doux (Boulineau et al., 2006). En outre, les oignons sont utilisés dans les produits destinés aux soins, à la beauté du visage et du corps, comme pommades, poudres et parfums. L’oignon a aussi des vertus en médecine, en réduisant le risque de maladies cardio-vasculaires (Craig, 1999). Certains composés de l’oignon comme les organo-sulfurés, flavonoïdes et fructanes sont conseillés pour assurer la santé du corps humain. Les composés organo-sulfurés donnent aux espèces du genre Allium leur gout et odeur spécifique (Kamenetsky et al., 2005).
3Pour conserver et utiliser la diversité génétique de l’oignon en Afrique, il faut d’abord la caractériser, c’est-à-dire mesurer son étendue et sa distribution. La présente étude a pour objectif de synthétiser les informations les plus pertinentes sur la biologie de l’oignon, sa position taxonomique, les ressources génétiques ainsi que les marqueurs morphologiques, biochimiques et moléculaires au service de l’analyse de la diversité génétique de l’oignon en Afrique. Les perspectives de cette étude doivent permettre de mettre en évidence des axes prioritaires de recherche pour mieux comprendre l’organisation de la variabilité génétique de l’oignon en Afrique et mieux gérer cette variabilité en termes de conservation et amélioration de l’espèce.
2. Biologie du développement et de la reproduction de l’oignon
4Le cycle cultural de l’oignon est généralement annuel pour la production des bulbes et bisannuel pour celle des graines (Fristsch et al., 2002). La tige de l’oignon est constituée par un plateau sur lequel s’insèrent des feuilles allongées, cylindriques et creuses et d’où partent des racines adventives. La base des feuilles peut se renfler lorsque les conditions sont favorables et former un bulbe surmonté par une fausse tige ou collet. Le bulbe d’oignon est composé d’écailles charnues et est recouvert extérieurement d’une ou de plusieurs couches d’écailles desséchées qui sont aussi appelées tuniques (Foury et al., 1992). La figure 1 illustre les différentes parties de la plante d’oignon.

5Après une phase de bulbification, puis d’arrêt de croissance et de dormance, le bulbe se remet normalement en végétation. Au cas où il y a initiation de la floraison, la plante peut émettre une ou plusieurs hampes florales. Ces dernières sont creuses, cylindriques, renflées en un endroit et se terminent par une ombelle composée de 200 à 700 fleurs bisexuées de couleur blanc verdâtre. Le fruit est une capsule contenant des graines de petite taille (200 à 300 par gramme), noires, anguleuses et dures. Chaque ombelle produit généralement 100 à 1 500 graines (De Lannoy, 2001).
6Chaque fleur a six étamines et un ovaire supère à trois loges contenant chacune deux gros ovules. Le pollen est émis avant que le stigmate ne soit réceptif. La fécondation croisée est donc dominante dans la mesure où les étamines sont mûres avant le pistil. Les abeilles (Apis mellifera L.), les bourdons (Bombus pascuorum Scopoli), les guêpes (Vespula vulgaris L.) et les mouches (Musca domestica L.) constituent les agents de pollinisation les plus actifs (De Lannoy, 2001 ; Fritsch et al., 2002). En Afrique, le regroupement des petites parcelles appartenant à plusieurs exploitants et la fécondation croisée de l’oignon favorisent des flux de gènes entre les écotypes cultivés (Rouamba et al., 1997). Cependant, une autofécondation est également possible entre les différentes fleurs d'une même plante, qui s'épanouissent successivement sur une même ombelle pendant une période de 3 à 4 semaines. Cette autofécondation, si elle est répétée, provoque une baisse de la vigueur des plantes obtenues à partir des graines (Foury et al., 1992).
7L'utilisation de la stérilité mâle est le moyen le plus économique pour produire commercialement des semences hybrides entre lignées sélectionnées. Jones et al. (1943) ont montré que la stérilité mâle de l’oignon est de type génocytoplasmique à déterminisme simple, avec un cytoplasme S (inducteur de stérilité) et des gènes nucléaires ms de maintien de la stérilité et Ms de restauration de la fertilité. Toutes les plantes avec le cytoplasme normal (N) sont fertiles, quel que soit le génotype au niveau du locus de restauration de la fertilité. Une deuxième source de stérilité mâle génocytoplasmique a été décrite chez des cultivars d'oignon créés en Europe (Berninger, 1965). Cette stérilité est déterminée par un cytoplasme T différent de S et trois gènes nucléaires récessifs, l’un étant indépendant des deux autres qui sont complémentaires (Schweisguth, 1973).
8Currah (2002) mentionne que le semis direct, le semis en pépinière suivi d’un repiquage et la plantation de bulbilles sont les trois modes d’installation de la culture observés dans les pays tropicaux d’Afrique. Toutefois, dans beaucoup de pays, notamment au Niger, le semis en pépinière est de loin la méthode de production la plus utilisée. Le semis des graines en pépinière, suivi d’un repiquage, permet le choix des plantes qui s’adaptent mieux à la concurrence au champ : on évite ainsi tout retard dans la reprise et la croissance ultérieure des jeunes plantes d’oignon.
9En Afrique, on assiste à une adoption partielle des semences certifiées de l’oignon produites par des centres de sélection variétale publics ou privés. En Afrique australe, les semences hybrides des variétés ‘ Tropicana F1 ’ et ‘ Dessex F1 ’ sont largement diffusées. Par contre, en Afrique de l’Ouest, les semences de la variété améliorée du Niger, ‘ Violet de Galmi ’, produites par une entreprise privée française, Technisem, constituent toujours le matériel le plus utilisé (Currah, 2002). Comme les producteurs de l’oignon du Niger maitrisent bien la production des semences, on constate que de nombreuses variétés paysannes de ce pays proviennent de l’autoproduction de semences paysannes à la ferme (Moumouni, 2006).
3. Ressources phytogénétiques
3.1. Origine et domestication
10L'oignon provient de la zone géographique comprenant la Turquie, l’Iran, l’Irak et le Pakistan (Hanelt, 1990). L’espèce A. cepa n’a pas été retrouvée à l’état spontané. Son parent le plus proche, A. vavilovii Popov & Vved., peut encore être observé à l’état spontané dans la région sise entre l'Iran, le Turkménistan et la Mongolie (Hanelt, 1990 ; Foury et al., 1992). Les traces des peintures sur les anciennes tombes égyptiennes témoignent que l'histoire de l’oignon remonte à au moins 3 200-2 800 avant Jésus-Christ. Ainsi, l’oignon était déjà une source de nourriture importante pour les habitants de l'Égypte ancienne (Boulineau et al., 2006). Selon Rouamba et al. (2001), les variétés de l’oignon d’Afrique tropicale ont pu être introduites à partir du sud de l’Égypte ou de l’Inde, via le Soudan, vers l’Afrique centrale et occidentale sous forme de graines ou de lots de bulbes génétiquement hétérogènes et ensuite sélectionnés par les agriculteurs locaux pour fournir des oignons mieux adaptés aux conditions écologiques de ces régions et des besoins des populations.
3.2. Position taxonomique
11La position taxonomique du genre Allium a fait l’objet de controverses. Dans la première classification des angiospermes, ce genre a été placé dans la famille des Liliaceae. Sur la base de la structure des inflorescences, Allium a été inclus dans les Amaryllidaceae. Cependant, avec l’avènement des marqueurs moléculaires, Allium est maintenant positionné au niveau d’une famille distincte, Alliaceae, proche de celle des Amaryllidaceae. Ainsi, le genre Allium appartient à la classe des Liliopsida, la sous-classe des Liliideae, le super ordre des Liliianae, l’ordre des Amaryllidales, la famille des Alliaceae, la sous-famille des Allioideae et la tribu des Allieae (Fristsch et al., 2002).
12Le genre Allium contient environ 780 espèces dont la majorité est présente dans l’hémisphère nord (Friesen et al., 2006). D’après Klaas et al. (2002), les espèces cultivées alimentaires ne représentent qu’une faible partie de la variabilité du genre. Friesen et al. (2006) classent les principales espèces cultivées du genre Allium dans les sous-genres Allium (ail, poireau) et Cepa (oignon, échalote, ciboule, ciboulette, ciboulette de Chine). Ce genre est divisé, soit en cinq sous-genres, en fonction des critères morphologiques et de la distribution géographique (Hanelt, 1990), soit en 15 sous-genres, en se basant sur les marqueurs moléculaires (Friesen et al., 2006).
13La section Cepa a été classée par Hanelt (1990) dans le sous-genre Rhizirideum (Koch). Toutefois, dans la dernière classification du genre Allium, Friesen et al. (2006) classent la section Cepa dans le nouveau sous-genre Cepa (Mill.) Radic.
14Gurushidze et al. (2007) mentionnent que la section Cepa (Mill.) Prokh. est constituée de douze espèces réparties en trois groupes d’espèces affines. Le premier groupe est composé des espèces A. cepa L., A. asarense R.M.Fritsch & Matin, A. farctum Wendelbo, A. roylei Stearn et A. vavilovii Popov & Vved. ; le second groupe est formé des espèces A. altaïcum Pall. et A. fistulosum L. ; le troisième groupe est constitué des espèces A. galanthum Kar. & Kir., A. oschaninii O.Fedtsch., A. praemixtum Vved. et A. pskemense B.Fedtsch.
3.3. Classification botanique et horticole
15En considérant les modes de propagation et de culture, Helm (1956) a classé A. cepa en quatre variétés botaniques : var. ‘ cepa ’ (oignon), var. ‘ viviparum ’ (oignon rocambole), var. ‘ aggregatum ’ (échalotes) et var. ‘ cepiforme ’ (petit oignon rouge de Chine ; ciboule).
16Plus tard, Jones et al. (1963) ont subdivisé l’espèce A. cepa en trois groupes horticoles : le groupe common onion, se caractérisant par des plantes produites par graines, donnant des inflorescences sans bulbilles et de gros bulbes normalement solitaires ; le groupe aggregatum, constitué d’échalotes à reproduction préférentiellement végétative et caractérisées par un bulbe souterrain semblable à celui de l'oignon, mais plus petit et divisé comme celui de l'ail et le groupe proliferum, caractérisé par des bulbes souterrains plus petits et des inflorescences portant des bulbilles qui assurent la multiplication.
3.4. Croisements interspécifiques
17Shigyo et al. (2008) mentionnent que les croisements naturels entre A. cepa et d’autres espèces du genre Allium sont rares et toujours stériles. En adoptant le concept de Harlan et al. (1971), le complexe d’espèces du genre Allium peut être structuré en trois pools géniques. Le pool primaire rassemble les quatre variétés botaniques de l’espèce A. cepa, intercompatibles (Helm, 1956). Le pool secondaire se compose des espèces qui peuvent s’hybrider avec l’oignon, mais le transfert de gènes par hybridation nécessite des techniques particulières pour surmonter des barrières d’incompatibilité. Dans le pool secondaire, les espèces A. altaicum, A. fistulosum, A. galanthum, A. roylei et A. vavilovii sont les plus exploitées, notamment pour améliorer la résistance de l’oignon aux maladies (Van Raamsdonk et al., 2003). Boulineau et al. (2006) indiquent que l’espèce A. roylei est employée comme « espèce pont » pour introduire chez A. cepa des gènes de résistances au mildiou (Peronospora destructor [Berkeley] Caspary) et à la pourriture du collet (Botrytis allii Munn) appartenant à l’espèce A. fistulosum. Le pool génétique tertiaire est constitué de toute espèce dont le transfert de gènes avec l’oignon se heurte à de très fortes barrières d’incompatibilité, nécessitant des techniques plus sophistiquées, comme l’hybridation somatique. Les espèces A. pskemense, A. oschaninii et une vingtaine d’espèces du sous-genre Cepa forment le pool tertiaire et peuvent être potentiellement exploitées pour l'amélioration de la résistance de l’oignon au mildiou, à la pourriture blanche (Sclerotium cepivorum Berk.) et à la pourriture du collet (Shigyo et al., 2008).
3.5. Collecte et conservation des ressources génétiques
18En Afrique, les ressources génétiques de l’oignon comprennent à la fois des variétés paysannes ou variétés populations et des variétés améliorées : les variétés paysannes ont été sélectionnées par les communautés rurales, tandis que les variétés améliorées ont fait l’objet de programmes de sélection génétique dans des centres de recherche (Grandval, 2011). Leland (1987) distingue aussi, au sein des variétés paysannes, des écotypes pour désigner des formes adaptées à une écologie bien spécifique, caractérisée par des facteurs biotiques et abiotiques particuliers.
19L’utilisation massive des variétés améliorées de l’oignon en Afrique fait craindre une baisse drastique de la diversité génétique, ce qui nécessite des mesures rapides de protection des ressources phytogénétiques de cette espèce (Rouamba et al., 2001). Sous l'égide de deux organisations, FAO à travers l’organisation RADHORT (Réseau Africain du Développement de l'Horticulture) et IPGRI, actuel Bioversity International, des réseaux ont été créés pour coordonner la collecte, la conservation et l'utilisation des espèces sauvages, des variétés améliorées, des variétés paysannes et des écotypes de l’oignon en Afrique (Currah, 2002). Dans cette partie du monde, il existe une très grande diversité de l’oignon, classée selon la couleur, la forme et la taille des bulbes, leur mode de culture, le gout, l’aptitude des bulbes à la conservation et la longueur du jour minimale nécessaire à la formation du bulbe. Le tableau 1 adapté à partir des données de Currah (2002), sur la base des rapports nationaux, donne les principales variétés paysannes et améliorées de l’oignon en Afrique.
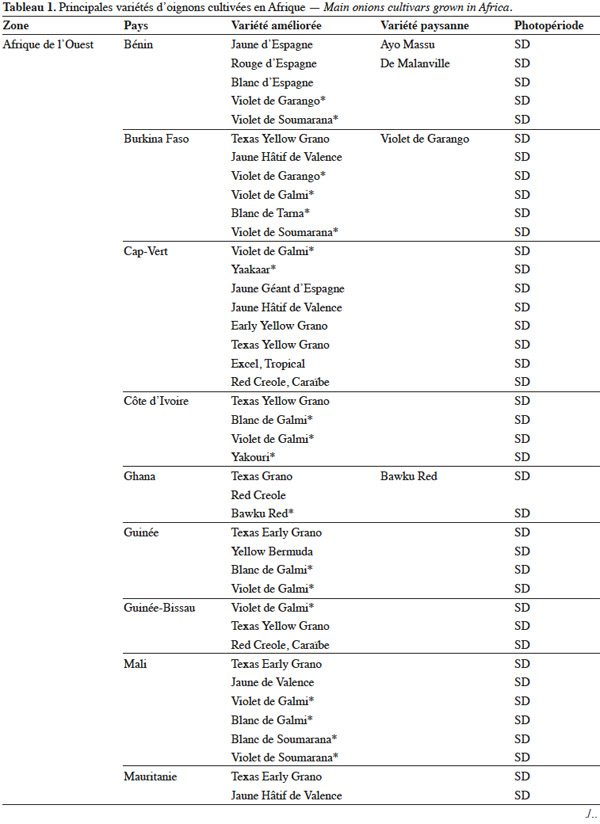
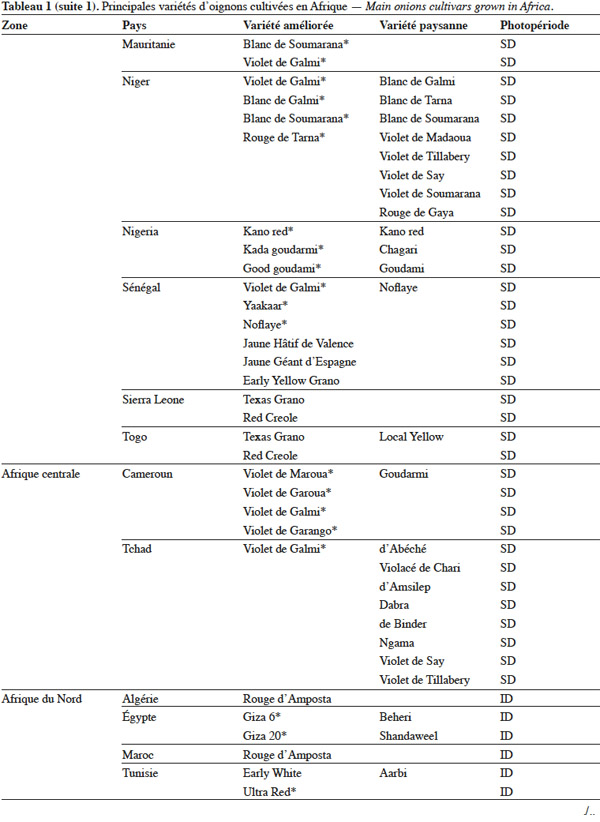
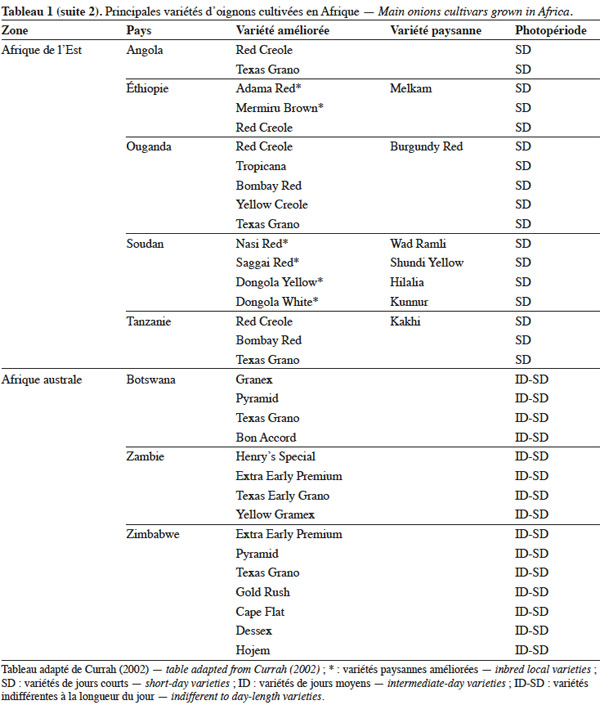
20En Afrique tropicale, une attention particulière a été accordée, d’une part, à la sélection de variétés adaptées aux conditions photopériodiques locales et, d’autre part, à l’introduction de variétés de jours courts originaires des États-Unis, des Caraïbes et de l’Inde, et de variétés de jours moyens originaires d’Europe. Nabos (1976) signale que les variétés de jours longs ne font pratiquement pas de bulbes en Afrique tropicale. Parmi les variétés sélectionnées en Afrique tropicale, De Lannoy (2001) et Currah (2002) citent, comme variétés de jours moyens, ‘ Jaunes Hâtif de Valence ’ et ‘ Jaune Géant d'Espagne ’ et comme variétés de jours courts, ‘ Early Texas Grano 502 PRR ’, ‘ Yellow Gramex ’, ‘ Red Creole ’, ‘ Violet de Galmi ’, ‘ Blanc de Galmi ’, ‘ Blanc de Soumarana ’ et ‘ Yaakaar ’.
21Au Niger, la sélection massale à partir des écotypes locaux ‘ Violet de Galmi ’, ‘ Blanc de Galmi ’ et ‘ Blanc de Soumarana ’ a permis de créer trois variétés améliorées qui portent respectivement les noms de ‘ IRAT 1 ’, ‘ IRAT 2 ’ et ‘ IRAT 3 ’. La variété ‘ IRAT 1 ’ qui porte son nom d’origine ‘ Violet de Galmi ’, est destinée à la consommation en frais, tandis que les deux autres variétés ‘ IRAT 2 ’ ou ‘ Blanc de Galmi ’ et ‘ IRAT 3 ’ ou ‘ Blanc de Soumarana ’, sont destinées à la déshydratation (Nabos, 1976). Une autre variété améliorée, ‘ Rouge de Tarna ’, a été créée à partir de la sélection d’un écotype local du Niger, ‘ Violet de Soumarana ’ (Rouamba et al., 2001).
22La cartographie des variétés paysannes et des variétés améliorées de l’oignon, réalisée par Rouamba et al. (1997) en Afrique de l’Ouest, a permis d’inventorier 83 variétés de l’oignon et 54 variétés de l’échalote, dans différentes zones de production de 13 pays. Les variétés les mieux adaptées aux conditions écologiques de l’Afrique de l’Ouest sont des variétés améliorées de jours courts, de couleur violette ou blanche, ‘ Violet de Galmi ’, ‘ Blanc de Galmi ’ et ‘ Blanc de Soumarana ’, originaires du Niger. On note cependant l’importance locale de variétés améliorées, comme ‘ Violet de Garango ’ au Burkina-Faso, ‘ Bawku Red ’ au Ghana, ‘ Rouge de Tarna ’ au Niger, ‘ Kano Red ’ au Nigeria et ‘ Noflaye ’ au Sénégal.
23Boukary et al. (2012), dans une étude sur l’interaction entre la variabilité des écotypes d’oignon et les facteurs agro-climatiques au Niger, ont identifié 21 écotypes. L’étude sur la taxonomie locale et l’identification des variétés paysannes de l’oignon par les producteurs du Niger a permis de montrer que 17 écotypes sont cultivés au Niger (Abdou et al., 2014). D’après Boukary et al. (2012), certains synonymes, par exemple les noms ‘ Violet de Galmi ’, ‘ Violet de Galmi Diffa ’, ‘ Violet de Galmi Gaya ’, ‘ Violet de Galmi Ayérou ’, ‘ Tassa ’, ‘ Kankaré ’ et ‘ Tawarka ’, désignaient des écotypes différents, mais les résultats des entretiens conduits par Abdou et al. (2015) montrent que des dénominations d’écotypes varient en fonction de la langue du producteur ; un même écotype peut avoir des noms vernaculaires différents d’un site à un autre.
24Pour comprendre la dynamique temporelle de perte et d’acquisition des écotypes d’oignon du Niger, la présence ou l’absence de chacun des écotypes identifiés à partir des recherches documentaires (Nabos, 1976 ; Rouamba et al., 1997 ; Rouamba et al., 2001 ; Silué et al., 2003 ; INRAN, 2004 ; Moumouni, 2006) a été comparée à la liste des écotypes identifiés par Abdou et al. (2014). La perte des écotypes ‘ Violet de Tahoua ’, ‘ Violet de Madaoua ’, des variétés améliorées ‘ Blanc de Galmi ’ et ‘ Blanc de Soumarana ’ est en partie justifiée par l’introduction de la variété améliorée ‘ Violet de Galmi ’. En outre, la prospection des nouveaux sites de production dans les régions de Dosso et de Zinder a permis d’identifier de nouveaux écotypes : ‘ Blanc de Soukoutantan ’, ‘ El Guidimouni ’, ‘ El Tassaou ’, ‘ El Gamdou ’.
25La caractérisation morphologique et l’évaluation agronomique des écotypes d’oignon identifiés par les producteurs, à l’aide des descripteurs scientifiques de International Plant Genetic Resources Institute, actuelle Bioversity International (IPGRI, 2001), a mis en évidence une diversité considérable entre et à l’intérieur des écotypes du Niger (Abdou et al., 2015).
26Concernant les échalotes, Currah (2002) indique que les variétés les plus cultivées en Guinée, en Côte d’Ivoire, au Mali, au Ghana, au Bénin et au Nigeria se caractérisent par des bulbes jaunes ou rouge-violet.
27En Afrique de l’Est et australe, les oignons sont représentés par des variétés originaires de l’Europe et des États-Unis : ce sont en général des variétés de jours moyens, bien adaptées aux conditions tropicales et subtropicales. Le Botswana, le Zimbabwe, la Zambie montrent une dominance des variétés à bulbes jaunes ‘ Yellow Gramex ’ et ‘ Yellow Creole ’. Au Kenya, en Tanzanie et en Ouganda, les variétés les plus cultivées sont des variétés importées, comme ‘ Red Creole ’ ou ‘ Rouge créole ’, ‘ Bombay Red ’ ou ‘ Rouge de Bombay ’, ‘ Texas Early Grano ’ et les variétés hybrides ‘ Tropicana F1 ’ et ‘ Dessex F1 ’. Dans les régions montagneuses d’Afrique orientale, les échalotes remplacent les oignons à cause du climat trop humide. Le cycle végétatif des échalotes est suffisamment bref pour permettre deux cycles de production par an.
28En Afrique du Nord, les variétés d’oignon les plus communément observées, ‘ Rouge d'Amposta ’ (Maroc et Algérie), ‘ Giza 6 ’ et ‘ Giza 20 ’ (Égypte), et ‘ Ultra Red ’ (Tunisie), sont de jours moyens et caractérisées par des bulbes de couleur rouge (Currah, 2002).
4. Marqueurs utilisés pour l’analyse de la diversité génétique
29Le tableau 1 montre qu’il existe une diversité de variétés de l’oignon en Afrique. Cependant, des variétés distinctes peuvent être inventoriées sous le même nom dans différents pays, comme une même variété peut avoir des noms différents suivant la région ou le pays. L’utilisation de marqueurs morphologiques, biochimiques et moléculaires contribue à mieux cerner l’organisation de la diversité génétique au sein de l’oignon en Afrique.
4.1. Marqueurs morphologiques
30Rouamba et al. (1997) signalent que la couleur et la forme des bulbes d’oignon sont les principaux descripteurs morphologiques qui permettent de différencier les variétés d’Afrique. Vingt-huit marqueurs morphologiques ont été identifiés à partir de caractères des bulbes, des feuilles, des tiges, des fleurs et des graines de l’oignon. Si ces marqueurs sont facilement observés à l’œil, ils ont l’inconvénient d’être dominants, d’être influencés par l’environnement et de dépendre souvent du stade de développement de la plante (Cramer et al., 1999). Le tableau 2 résume les principaux marqueurs qualitatifs de l’oignon et leurs déterminismes génétiques.
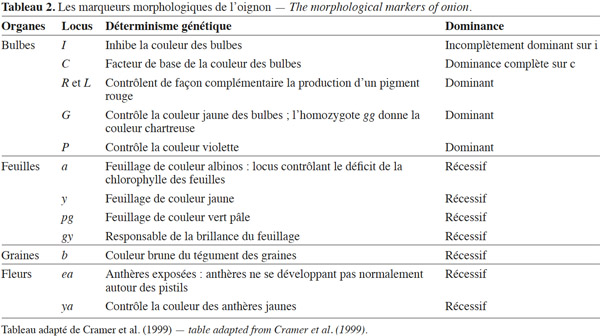
31Descripteurs des bulbes. Les bulbes des variétés de l’oignon diffèrent considérablement par leur forme sphérique, aplatie, conique, allongée ; leur couleur ; leur gout et leur aptitude à la conservation (Shigyo et al., 2008).
32Blanche, jaune, brune, rouge ou violette, la couleur des bulbes a été utilisée comme un critère majeur pour analyser la diversité génétique, ainsi que pour classer, sélectionner et créer de nouvelles variétés de l’oignon (Kim et al., 2009). Les oignons d’Afrique de l’Ouest sont de couleur violette, blanche et parfois jaune, alors que les autres pays d’Afrique montrent une dominance des variétés à bulbes rouges ou jaunes (Currah, 2002).
33La couleur est principalement régie par une série de gènes à hérédité mendélienne mono- et oligogénique (Reiman, 1931). La couleur blanche du bulbe peut être attribuée soit à un gène inhibiteur (II) de couleur incomplètement dominant, qui supprime toute coloration, soit à un gène récessif (rr) conduisant aux mutants incolores apparus dans les variétés de couleur rouge, jaune ou brune (Davis et al., 1967). Ces mutants ont servi de point de départ à la création de nouvelles variétés de l'oignon à bulbe blanc au Niger (Nabos, 1976). Kim et al. (2004) indiquent l’existence d’un autre allèle (P) indépendant qui contrôle la couleur violette des bulbes. Selon Fossen et al. (1996), la présence des composés flavonoïdes de la famille des anthocyanes produit au niveau du bulbe des couleurs variant du rouge au violet.
34Descripteurs des feuilles. Les pigments chlorophylliens des feuilles de l’oignon sont variables et permettent d’identifier les types d’oignon. La couleur des feuilles des plantules de l’oignon peut être blanche, jaune, verte pâle ou verte. Jones et al. (1944) ont étudié le déterminisme génétique du déficit de la chlorophylle des feuilles de l’oignon. Les plantules albinos (a), jaunes (y), vertes pâles (pg) ont toutes des génotypes homozygotes récessifs et meurent immédiatement après la germination. Selon les mêmes auteurs, la brillance du feuillage, résultant de la présence de cire sur la surface des feuilles, est conditionnée par des allèles récessifs au locus (gy).
35Descripteurs des fleurs. Plusieurs loci régissent la stérilité mâle et la morphologie florale de l'oignon. Chez certaines plantes, les périanthes ne se développent pas normalement autour des anthères. Davis (1966) mentionne que ces traits font référence aux anthères exposées ou « exposed anthers » (ea) et sont conditionnés par les génotypes récessifs à ce locus (ea). Selon Jones et al. (1944), la couleur des anthères est contrôlée par le locus ya (yellow anthers) : ya ya correspond aux plantes à anthères jaunes et Ya Ya ou Ya ya correspondent aux plantes à anthères vertes. En outre, le périanthe blanc a été signalé comme étant récessif et contrôlé par un seul locus (Davis, 1966).
36Descripteurs des graines. Les différentes variétés de l’oignon ont des graines à tégument noir ou brun. Selon Davis (1966), la couleur du tégument des graines est déterminée par le locus b avec B_ donnant un tégument noir et le génotype bb donnant un tégument brun.
4.2. Marqueurs biochimiques
37Selon Cramer et al. (1999), 24 enzymes différentes ont été caractérisées dans les graines et les racines pour analyser la diversité entre l’espèce A. cepa et les autres espèces du genre Allium. L’analyse de la diversité génétique dans une collection de 188 variétés améliorées de l’oignon originaires des États-Unis à l’aide des enzymes alcool déshydrogénase (ADH), isocitrate déshydrogénase (IDH), phosphoglucomutase (PGM), phospholglucoisomerase (PGI) a permis de montrer que seule l’enzyme alcool déshydrogénase est polymorphe entre les variétés de l’oignon (Peffley et al., 1987). Cependant, Rouamba et al. (2001) signalent que les enzymes alcool déshydrogénase (ADH), 6-phosphogluconate déshydrogénase (6-PGDH), estérase (EST), phosphoglucomutase (PGM), phospholglucoisomerase (PGI) et malate déshydrogénase (MDH) sont polymorphes au sein de 16 écotypes d’oignon originaires de six pays d’Afrique de l’Ouest.
38D’une manière générale, l’ensemble des systèmes enzymatiques chez l’oignon peut être réparti en deux groupes. Un premier groupe constitué des enzymes ADH, MDH, 6-PGDH, PGM, EST et PGI, dont la structure et l’hérédité sont connues. Toutefois, la structure et l’hérédité des systèmes PGI et MDH sont controversées. Selon Peffley et al. (1987), la PGI et la MDH seraient codées chacune par deux loci. Par contre, Rouamba et al. (2006) montrent que l’enzyme PGI est codée par un locus avec trois allèles, la MDH serait codée par trois loci dont deux seraient monomorphes et un polymorphe. Le deuxième groupe est constitué de toutes les autres enzymes dont la structure et l’hérédité ne sont pas encore connues.
39L’évaluation de la diversité génétique des écotypes d’oignon provenant de l’Afrique de l’Ouest, à l’aide de quatre enzymes (ADH, 6-PGDH, PGI et MDH), a permis à Rouamba et al. (1997) de séparer des variétés provenant des pays francophones : Bénin, Burkina Faso, Côte d’ivoire, Mali, Niger, des variétés du Nigeria, anglophone. Ces auteurs suggèrent l’absence de flux de gènes entre ces cinq pays francophones et le Nigeria. En général, l’analyse de la diversité génétique de l’oignon d’Afrique à l’aide de marqueurs enzymatiques montre une homogénéité biochimique entre plusieurs variétés ou entre individus d’une même variété, qui ne se reflète pourtant pas au niveau phénotypique, en particulier pour la forme et la couleur des bulbes (Rouamba et al., 2001).
40Par ailleurs, le polymorphisme des enzymes estérase, alcool déshydrogénase, isocitrate déshydrogénase, phospholglucoisomerase a été utilisé pour distinguer le profil génétique de l’oignon, de la ciboule et les hybrides des deux espèces (Cryder et al., 1991 ; Peffley et al., 2000).
4.3. Marqueurs moléculaires
41À notre connaissance, aucune étude n’a été faite pour analyser la diversité génétique des variétés de l’oignon d’Afrique à l’aide des marqueurs moléculaires. Pourtant, les marqueurs Random Amplified Polymorphic DNA (RAPD), Restriction Fragment Length Polymorphism (RFLP), Amplified Fragment Length Polymorphism (AFLP), Target Region Amplification Polymorphism (TRAP) et Simple Sequence Repeats (SSRs) ont été utilisés avec succès pour refléter des variations organoleptique et morphologique entre différentes variétés de l’oignon, ainsi qu’entre l’oignon et d’autres espèces du genre Allium (Klaas et al., 2002).
42Wilkie et al. (1993) ont utilisé la technique moléculaire RAPD avec 20 amorces aléatoires pour analyser des variations entre six variétés de l'oignon dont cinq de jours longs et une de jours courts. Parmi les 20 amorces, sept ont révélé des polymorphismes entre les variétés de l’oignon, mais ces marqueurs RAPD ne permettent cependant pas de discriminer les variétés de l’oignon en fonction de la sensibilité à la photopériode.
43La technique RFLP a permis d’observer une différenciation génétique entre, d’une part, les variétés améliorées de l’oignon de jours longs originaires de l’Europe (Espagne et Hollande) et, d’autre part, celles de jours courts originaires des États-Unis (King et al., 1998).
44Les marqueurs dominants AFLP ont été peu utilisés pour l’analyse de la diversité génétique de l’oignon à cause de la taille du génome nucléaire, du nombre élevé d’hétérozygotes et du faible taux de la diversité allélique (Van Raamsdonk et al., 2003). D’après King et al. (1998), les marqueurs AFLP ont révélé une absence de polymorphisme chez les variétés hybrides et les variétés paysannes de l’oignon originaires d’Europe, du Nord des États-Unis et du Japon.
45Fischer et al. (2000) ont été les premiers à développer 30 marqueurs microsatellites génomiques (SSRg) qui ont permis d’analyser la diversité entre 83 variétés de l’oignon venant d’une vingtaine de pays d’Europe, d’Amérique et d’Asie. Cependant, seuls quatre marqueurs sont polymorphes, ce qui ne permet pas la discrimination inter-variétale. Plus tard, Kuhl et al. (2004), Martin et al. (2005), McCallum et al. (2008), Kharl et al. (2010) et Baldwin et al. (2012) ont identifié des marqueurs SSRg et SSRest (Expressed Sequence Tag): ceux-ci ont conduit non seulement à la discrimination entre les variétés de l’oignon, mais aussi à mieux caractériser la diversité génétique intra- et inter-variétale. En effet, l’utilisation de 20 marqueurs SSRg a permis à Baldwin et al. (2012) de regrouper, notamment en fonction de la sensibilité à la photopériode, 24 variétés d’oignon originaires d’Amérique du Sud, d’Espagne, du Portugal, d’Inde et de Nouvelle Zélande.
5. Conclusions et recommandations
46L’oignon est une plante herbacée bisannuelle de la famille des Alliaceae, cultivé pour l’alimentation humaine depuis des milliers d'années dans les régions tempérées et subtropicales. Malgré l’identification d’un grand nombre de variétés et l’évolution des techniques d’analyse de la diversité génétique, très peu d’études ont été menées sur la diversité intra- et inter-variétale de l’oignon en Afrique. Tenant compte des données des rapports nationaux d’inventaires des variétés paysannes et améliorées de l’oignon, il nous semble que le nombre de variétés d’oignons répertoriées en Afrique ne correspond pas à la réalité. Les variétés originaires d’un même pays sont cultivées dans différents pays. Par exemple, les variétés améliorées du Niger sont rencontrées dans tous les pays de l’Afrique de l’Ouest. Il ressort aussi de cette étude bibliographique que les variétés paysannes de l’oignon d’Afrique sont menacées de disparition face à l’introduction massive des variétés améliorées importées des États-Unis, d’Europe, du Japon. Pour conserver ces variétés paysannes, il faut les identifier, les caractériser et les évaluer.
47Il est essentiel d’analyser les flux de gènes au niveau intra- et inter-variétal, grâce à l’utilisation des marqueurs moléculaires microsatellites et d’évaluer l’impact de ces flux de gènes dans des programmes de conservation in situ et ex situ, ce qui permettrait de concevoir les meilleures stratégies de conservation génétique de l’espèce en Afrique. La richesse des variétés de cette espèce doit être étudiée avec l’appui de divers outils d’identification : ethnologique, morphologique, agronomique, biochimique et moléculaire. Ces résultats pourraient contribuer à mieux comprendre, non seulement le rôle des producteurs dans la gestion de la diversité génétique, mais aussi l’organisation de cette diversité dans une perspective d’amélioration des types locaux.
Bibliographie
Abdou R. et al., 2014. Taxonomie locale et analyse des critères des paysans pour caractériser les différents écotypes d'oignon (Allium cepa L.) du Niger. Cah. Agric., 23, 166-176.
Abdou R. et al., 2015. Variabilité morphologique et agronomique des écotypes d’oignon (Allium cepa L.) identifiés par les producteurs du Niger. Tropicultura, 33, 1-14.
Baldwin S. et al., 2012. Development of robust genomic simple sequence repeat markers for estimation of genetic diversity within and among bulb onion (Allium cepa L.) populations. Mol. Breeding, 30, 1401-1411.
Berninger E., 1965. Contribution à l’étude de la stérilité mâle de l’oignon (Allium cepa L.). Ann. Amelior. Plantes, 15, 183-199.
Boukary H. et al., 2012. Interactions entre la variabilité des écotypes de l’oignon (Allium cepa L.) et les facteurs agro-climatiques au Niger. Tropicultura, 30(4), 209-215.
Boulineau F. et al., 2006. L’oignon. In : Doré C. & Varoquaux F., éds. Histoire et amélioration de cinquante plantes cultivées. Paris : INRA, 481-493.
Craig W.J., 1999. Health-promoting properties of common herbs. Am. J. Clin. Nutr., 70(3), 491-499.
Cramer C.S. & Havey M.J., 1999. Morphological, biochemical, and molecular markers in onion. Hortic. Sci., 34, 589-593.
Cryder C.M., Corgan J.N., Urquhart N.S. & Clason D., 1991. Isozyme analysis of progeny derived from (Allium fistulosum × Allium cepa) × Allium cepa. Theor. Appl. Genet., 82, 337-345.
Currah L., 2002. Onions in the tropics: cultivars and country reports. In : Rabinowitch H.D. & Currah L., eds. Allium crop science: recent advances. Wallingford, UK; New York, USA: CABI Publishing, 379-408.
Davis E.W., 1966. Marker genes to facilitate roguing onion-seed fields. Seed World, 87, 4-6.
Davis G.N. & El-Shafie M.W., 1967. Inheritance of bulb color in the onion (Allium cepa L.). Hilgardia, 38, 607-622.
De Lannoy G., 2001. Oignon Allium cepa L. In : Raemaekers R.H., ed. Agriculture en Afrique tropicale. Bruxelles : DGCI, 518-526.
FAOSTAT, 2013. Base de données statistiques agricoles FAO, http://faostat3.fao.org/browse/rankings/countries_by_commodity/F, (12/05/2015).
Fischer D. & Bachmann K., 2000. Onion microsatellites for germplasm analysis and their use in assessing intra- and interspecific relatedness within the subgenus Rhizirideum. Theor. Appl. Genet., 101, 153-164.
Fossen T. et al., 1996. Characteristic anthocyanin pattern from onions and other Allium spp. J. Food Sci., 61, 703-706.
Foury C. & Schweisguth B., 1992. L’oignon. In : Gallais A. & Bannerot H., éds. Amélioration des espèces végétales cultivées. Paris : INRA, 406-419.
Friesen N., Fritsch R.M. & Blattner F.R., 2006. Phylogeny and new intrageneric classification of Allium L. (Alliaceae) based on nuclear ribosomal DNA ITS sequences. Aliso, 22, 372-395.
Fritsch R.M. & Friesen N., 2002. Evolution, domestication and taxonomy. In: Rabinowitch H.D. & Currah L., eds. Allium crop science: recent advances. Wallingford, UK; New York, USA: CABI Publishing, 5-30.
Grandval F., 2011. Quelques définitions clés pour aborder ce dossier « semences ». Grain Sel, 52-53, 39-40.
Gurushidze M. et al., 2007. Phylogenetic relationships of wild and cultivated species of Allium section Cepa inferred by nuclear rDNA ITS sequence analysis. Plant Syst. Evol., 269, 259-269.
Hanelt P., 1990. Taxonomy evolution and history. In: Rabinowitch H.D. & Brewster J.L., eds. Onions and allied crops. Boca Raton, FL, USA: CRC Press Inc, 1-26.
Harlan J.R. & de Wet J.M.J., 1971. The origin and domestication of Sorghum bicolor. Econ. Bot., 25, 128-135.
Helm J., 1956. Die zu Würz-und Speisezwecken kultivierten Arten der Gattung Allium L. Kulturpflanze, 4, 130-180.
INRAN, 2004. Rapport d'activité, collecte et épuration des cultivars locaux d'oignon. Rapport d'activité de la campagne 2002-2003. Niamey : PPEAP & INRAN.
IPGRI, ECP/GR, AVRDC, 2001. Descriptors for Allium (Allium spp.). Roma: International Plant Genetic Resources Institute; Roma: European Cooperative Programme for Crop Genetic Resources Networks; Tainan, Taiwan: Asian Vegetable Research and Development Center.
Jones H.A. & Clarke A.E., 1943. Inheritance of male sterility in the onion and the production of hybrid seed. Proc. Am. Soc. Hortic. Sci., 43, 189-194.
Jones H.A., Clarke A.E. & Stevenson F.J., 1944. Studies in the genetics of the onion (Allium cepa L.). Proc. Am. Soc. Hortic. Sci., 44, 479-484.
Jones H.A. & Mann L.K., 1963. Onions and their allies. Botany, cultivation and utilization. New York, USA: Interscience.
Kamenetsky R. et al., 2005. Diversity in fertility potential and organo-sulphur compounds among garlics from Central Asia. Biodivers. Conserv., 14, 281-295.
Kharl A., Lawande K.E. & Negi K.S., 2010. Microsatellite marker based analysis of genetic diversity in short day tropical Indian onion and cross amplification in related Allium spp. Genet. Resour. Crop Evol., 58, 741-754.
Kim S. et al., 2004. Pink (P), a new locus responsible for a pink trait in onions (Allium cepa) resulting from natural mutations of anthocyanidin synthase. Mol. Genet. Genomics, 272, 18-27.
Kim S. et al., 2009. Identification of two novel inactive DFR-A alleles responsible for failure to produce anthocyanin and development of a simple PCR-based molecular marker for bulb color selection in onion (Allium cepa L.). Theor. Appl. Genet., 118, 1391-1399.
King J.J., Bradeen J.M. & Havey M.J., 1998. Variability for restriction fragment-length polymorphisms (RFLPs) and relationships among elite commercial inbred and virtual hybrid onion populations. J. Am. Soc. Hortic. Sci., 123, 1034-1037.
Klaas M. & Friesen N., 2002. Molecular markers in Allium. In: Rabinowitch H.D. & Currah L., eds. Allium crop science: recent advances. Wallingford, UK; New York, USA: CABI Publishing, 159-186.
Kuhl J.C. et al., 2004. A unique set of 11,008 onion expressed sequence tags reveals expressed sequence and genomic differences between the monocot orders Asparagales and Poales. Plant Cell, 16, 114-125.
Leland R.H., 1987. Manuel pour la sélection du sorgho. 2e éd. Andhra Pradesh, Inde : ICRISAT.
Martin W.J. et al., 2005. Genetic mapping of expressed sequences in onion and in silico comparisons with rice show scant colinearity. Mol. Genet. Genomics, 274, 197-204.
McCallum J. et al., 2008. Genetic diversity analysis and single-nucleotide polymorphism marker development in cultivated bulb onion based on expressed sequence tag-simple sequence repeat markers. J. Am. Soc. Hortic. Sci., 133, 810-818.
Moumouni A.D., 2006. Les effets de la réappropriation de la culture du « Violet de Galmi » par les producteurs d’oignon de la région de Tahoua – NIGER, sur la dynamique du territoire local, l’organisation sociale et économique. Thèse de doctorat : Université de Toulouse - Le Mirail (France).
Nabos J., 1976. L’amélioration de l’oignon (Allium cepa L.) au Niger. Agron. Trop., 31(4), 387-397.
Peffley E.B. & Orozco-Castillo C., 1987. Polymorphism of isozymes within plant introductions of A. cepa L. and A. fistulosum L. HortScience, 22, 956-957.
Peffley E.B. & Hou A., 2000. Bulb-type onion introgressants posessing Allium fistulosum L. genes recovered from interspecific hybrid backcrosses between A. cepa L. and A. fistulosum L. Theor. Appl. Genet., 100(3-4), 528-534.
Reiman H., 1931 Genetic factors for pigmentation in the onion and their relation to disease resistance. J. Agric. Res., 42, 251-278.
Rouamba A., Sarr A. & Ricroch A., 1997. Dynamic management of genetic ressource of Allium cepa L. (onion) in west Africa. Acta Hortic., 433, 185-189.
Rouamba A., Sandmeier M., Sarr A. & Ricroch A., 2001. Allozyme variation within and among populations of onion (Allium cepa L.) from West Africa. Theor. Appl. Genet., 103, 855-861.
Rouamba A. & Ricroch A., 2006. La phosphoglucoisomérase et la malate deshydrogénase : deux nouveaux systèmes enzymatiques polymorphes chez l'oignon. Agron. Afr., 18(2), 135-144.
Schweisguth B., 1973. Étude d’un nouveau type de stérilité mâle chez l’oignon, Allium cepa L. Ann. Amelior. Plantes, 23, 221-233.
Shigyo M. & Kik C., 2008. Onion. In: Prohens J. & Nuez F., eds. Vegetables II: Fabaceae, Liliaceae, Solanaceae, and Umbelliferae. New York, USA: Springer, 121-159.
Silué S., Fondio L., Coulibaly M.Y. & Magein H., 2003. Sélection de variétés d’oignon (Allium cepa L.) adaptées au nord de la Côte d’Ivoire. Tropicultura, 21(3), 129-134.
Van Raamsdonk L.W.D. et al., 2003. Biodiversity assessment based on cpDNA and crossability analysis in selected species of Allium subgenus Rhizirideum. Theor. Appl. Genet., 107, 1048-1058.
Wilkie S.E., Isaac P.G. & Slater R.J., 1993. Random amplified polymorphic DNA (RAPD) markers for genetic analysis in Allium. Theor. Appl. Genet., 86, 497-504.