- Accueil
- volume 10 (2006)
- numéro 3
- Etude par les isozymes de la pollinisation libre de génotypes d’hévéas sauvages (Hevea brasiliensis Muell. Arg.) en verger à graines
Visualisation(s): 0 (0 ULiège)
Téléchargement(s): 0 (0 ULiège)
Etude par les isozymes de la pollinisation libre de génotypes d’hévéas sauvages (Hevea brasiliensis Muell. Arg.) en verger à graines

Notes de la rédaction
Reçu le 6 septembre 2005, accepté le 5 avril 2006
Résumé
La Côte d’Ivoire dispose d’une population sauvage d’hévéas pour laquelle une amélioration en verger à graines est envisagée par pollinisation libre. L’étude de la pollinisation libre de génotypes sauvages d’hévéas, plantés en verger à graines par 5 loci isozymes, a confirmé le caractère allogame de l’hévéa. Sur la période de l’étude (1998 et 1999), le taux d’allofécondation estimé à 0,97 et 0,96 est peu variable d’une année à l’autre. L’importance du taux d’allofécondation montre que les croisements se font majoritairement entre arbres de génotypes différents. L’index de fixation, ou FIS, diminue fortement de la population parentale à celle des descendants où il n’est pas significativement différent de zéro. Les croisements se font de façon panmictique entre les arbres de l’essai et les fécondations entre les arbres apparentés sont pratiquement inexistantes. Le taux d’autofécondation est majoritairement dû aux autofécondations réelles entre les ovules et le pollen provenant de mêmes origines. Ainsi, compte tenu des résultats obtenus, la pollinisation entre les arbres de l’essai serait favorable à la réalisation de gains génétiques substantiels dans la population d’hévéas sauvages.
Abstract
Survey of the open pollination by isozymes in a wild rubber trees (Hevea brasiliensis Muell. Arg.) seeds orchard. The Côte d’Ivoire has a wild rubber tree population for which an improvement in a seeds orchard is suggested by open pollination. The isozymes markers (5 loci) were applied to study the open pollination in a rubber tree seed orchard in 1998 and 1999. The results confirmed the allogamic character of the rubber tree. On the period of the survey (1998 and 1999), the outcrossing rate estimated to 0.97 and 0.96 varies weakly between the two years. The high level of the outcrossing rate show that pollinations are realized between trees of different genotypes. The fixation index (FIS) decrease from the parental population to the descendant’s one where it is not significantly different from zero. So crossings are panmictics, and the fertilizations between the related trees are practically inexistent. The rate of self-fertilization is mostly due to the real self-fertilizations between the ovuls and the pollen coming from the same origin. Thus, considering the results, the pollination in the seeds orchard would be favorable to the realization of substantial genetic progress in the wild rubber trees population.
Table des matières
1. Introduction
1Hevea brasiliensis Muell. Arg. (ou hévéa) est une espèce forestière tropicale à pollinisation entomophile (Rao, 1961) et productrice de caoutchouc qui est utilisé principalement dans la fabrication des pneumatiques. L’amélioration génétique de l’hévéa est confrontée à plusieurs problèmes dont le plus important est le faible taux de réussite à la pollinisation manuelle représentant le seul moyen d’obtention d’hybrides légitimes. En Côte d’Ivoire, ce taux de réussite à la pollinisation manuelle est évalué à 3 % (Nicolas, 1979 ; Leconte, 1984). Chaque année, près de 30000 pollinisations sont réalisées pour obtenir 2000 hybrides constituant la structure d’entrée dans un schéma de sélection à trois étapes (Legnaté, Clément-Démange, 1991) : le Champ d’Evaluation de Seedling (semenceaux) ou CES ; le Champ de Clones à Petite Echelle ou CCPE ; le Champ de Clones à Grande Echelle ou CCGE.
2Ces pollinisations manuelles représentent une masse importante de travail limitant l’évaluation en croisement de tous les géniteurs disponibles en collection. Ainsi, l’utilisation dans le schéma d’amélioration de 3000 génotypes sauvages introduits en Côte d’Ivoire en 1981 (Nicolas, 1981), s’avère pratiquement impossible à l’échelle humaine par l’utilisation de la pollinisation manuelle. Aussi, compte tenu du faible niveau de production de ces génotypes sauvages (Clément-Démange et al., 1995), l’utilisation de la pollinisation libre est-elle envisagée dans un schéma de sélection récurrente, pour les évaluer avant de les croiser avec les meilleurs génotypes cultivés. Cette évaluation se ferait entre les groupes génétiques d’hévéas sauvages définis (Chevallier, Lebrun, 1988), alternant des cycles de recombinaison par pollinisation libre en verger à graines et d’évaluation par sélection précoce de leurs descendants (Baudoin et al., 1997). Les génotypes présentant les meilleures aptitudes générales à la combinaison seraient retenus en fin de sélection. L’application d’une telle méthodologie de sélection suppose que le mode de reproduction en pollinisation libre, c’est-à-dire le mode de brassage des gènes, est connu et maîtrisé ce qui n’est pas le cas chez l’hévéa. Le présent travail a été réalisé sur deux années successives, en 1998 et en 1999, pour étudier le mode de reproduction en pollinisation libre de l’hévéa afin de juger a priori de l’efficacité d’une telle méthode de sélection.
2. Matériel et Méthodes
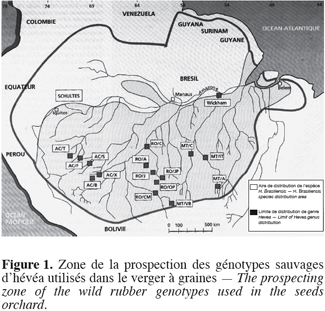
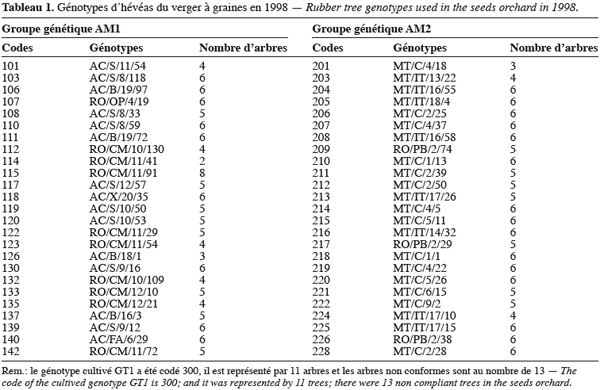
3Le matériel utilisé est constitué de 50 génotypes sauvages et d’un génotype cultivé mâle stérile (Leconte, 1984) appelé GT1. Les génotypes sauvages ont été prospectés dans trois états brésiliens (Nicolas, 1981) : l’Acre, le Rondonia et le Mato-Grosso (Figure 1). Ils sont répartis équitablement entre deux groupes génétiques AM1 et AM2 (Tableau 1) mis en évidence par la structuration de leur variabilité génétique par l’utilisation des isozymes (Chevallier, 1988). Le groupe AM1 est constitué des génotypes de l’Acre et d’une grande partie des génotypes du Rondonia. Quant au groupe AM2, il est majoritairement constitué des génotypes du Mato-Grosso.
4Tous ces génotypes ont été greffés dans un verger qui a été établi sur la station expérimentale de cacao et de café du CNRA (Centre National de Recherche Agronomique) de Divo (200 km au Nord d’Abidjan). Ce site est très éloigné de toute plantation d’hévéa afin d’éviter des pollinisations extérieures.
5Les génotypes étaient représentés par 4 à 8 individus pour les génotypes sauvages et par 11 individus pour le génotype cultivé sur une superficie de 0,89 ha. La densité de plantation est de 330 arbres à l’hectare soit 312 arbres pour la superficie de l’essai. Six mètres séparent 2 arbres sur une même ligne et la distance entre 2 lignes mitoyennes est de 5,20 m. Les différents individus des génotypes étudiés ont été disposés aléatoirement sur toute la superficie de l’essai. Excepté les individus de bordures, chaque arbre se retrouve au milieu d’un hexagone régulier de 6 m de côté dont les sommets sont occupés dans la plupart des cas, par des individus de différents génotypes. Cette disposition permet a priori d’éviter les autofécondations entre arbres de même génotypes.
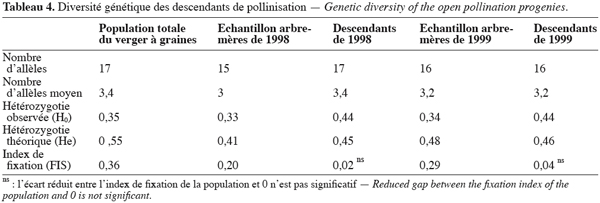
6A la mise en place, le verger à graines avait un peuplement de 312 arbres qui a diminué au fil du temps à cause des déracinements et de la verse due au vent. Ainsi, en 1998, l’essai comportait 287 arbres et, en 1999, 286 arbres.
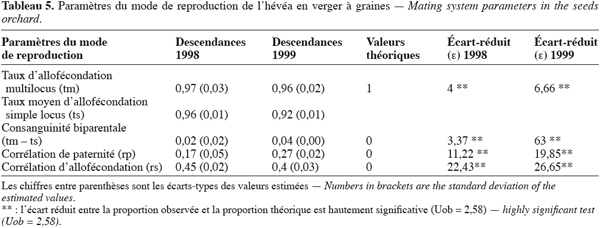
7Tous les arbres du verger à graines ont été analysés par isozymes ainsi que 2 échantillons, l’un de 615 en 1998 et l’autre de 1065 en 1999 (Tableau 2). Les échantillons de descendants ont été prélevés sur 29 arbres de 15 génotypes en 1998 et sur 39 arbres de 17 génotypes en 1999. Les analyses isoenzymatiques ont été réalisées à partir de feuilles fraîches anthocyanées, à pH 6 sur un gel d’amidon en utilisant 5 systèmes isoenzymatiques qui sont :
8– la phosphoglucoisomérase ou PGI (E.C. : 5.1.3.9) ;
9– la phosphatase acide ou PAC (E.C. : 3.1.3.2.) ;
10– la leucine amino peptidase ou LAP (E.C. : 3.4.11.1.) ;
11– l’alcool déshydrogénase ou ADH (E.C. : 1.1.1.1.) ;
12– l’estérase ou EST (E.C. : 3.1.1.1).

13Avant l’étude du mode de reproduction, la conformité entre les arbres de même génotype a été étudiée en comparant les zymogrammes des individus de même génotype (ramet). De même, la diversité génétique des géniteurs sauvages et de leurs descendants issus de pollinisation libre a été étudiée à partir des données enzymatiques, par l’estimation des paramètres suivants : le nombre total d’allèles dans la population étudiée qui est la somme des allèles aux loci considérés ; le nombre moyen d’allèles par locus ; les fréquences alléliques c’est-à-dire le rapport entre le nombre de copies de chaque allèle dans la population et le nombre total de copies de tous les allèles ; l’hétérozygotie observée Ho ou la fréquence d’apparition d’hétérozygotes par locus dans la population ; l’hétérozygotie théorique He sous l’hypothèse de la panmixie (croisement aléatoire) ou indice de diversité de Nei (Nei, 1987) ; l’hétérozygotie observée Ho et l’hétérozygotie attendue He ont permis de déterminer l’index de fixation de Wright (Wright, 1964) ou FIS qui mesure la réduction de l’hétérozygotie attendue sous hypothèse de la panmixie : FIS = 1-Ho/He. C’est également une mesure du taux de la consanguinité de la population (Demarly, 1977).
14Les paramètres du mode de reproduction ont ensuite été estimés par le maximum de vraissemblance (Ritland, El-Kassaby, 1985 ; Ritland, 1994) à partir du modèle à fécondation mixte (Ritland, Jain, 1981).
15Ce modèle suppose qu’une partie (t) des graines d’un arbre provient d’allofécondations et qu’une autre partie (1-t) est issue d’autofécondations. Les paramètres estimés sont : le taux d’allofécondation multilocus ou tm qui est la proportion de descendants issus de la rencontre de gamètes d’origines génétiques différentes décelée par utilisation de tous les loci ; le taux d’allofécondation moyen simple locus ou ts ; la consanguinité biparentale tm-ts qui est la composante du taux d’autofécondation attribuée aux croisements entre apparentés. Dans le verger à graines, on considérera les arbres de même génotype comme individus apparentés ; la corrélation d’allofécondation rs, qui indique la variation du taux d’allofécondation dans les descendances ; la corrélation de paternité rp, ou proportion de pleins-frères parmi les descendants issus d’allofécondation.
16Les valeurs de ces paramètres ont été comparées aux valeurs théoriques 0 ou 1 par le test de l’écart réduit (Dagnelie, 1998). Pour l’estimation de ces différents paramètres chaque arbre a été considéré comme un individu différent dans un même ramet.
3. Résultats
3.1. Conformité clonale dans le verger à graines
17La vérification de la conformité clonale entre les individus de même génotype à partir de leur zymogramme a montré que 13 arbres ne sont pas conformes, à leur présumé génotype. Ces arbres non conformes, qui représentent un taux d’erreur de 4,5 %, pourraient provenir du développement d’un bourgeon du porte-greffe en lieu et place du greffon. Il pourrait également s’agir d’erreurs de greffage, c’est-à-dire du greffage d’un génotype inconnu en lieu et place d’un génotype à greffer.
3.2. Diversité génétique des géniteurs et de leurs descendants
18Dans la population de géniteurs du verger à graines, les 5 loci isoenzymatiques utilisés forment un total de 17 allèles soit un nombre moyen de 3,4 allèles par locus (Tableau 3).

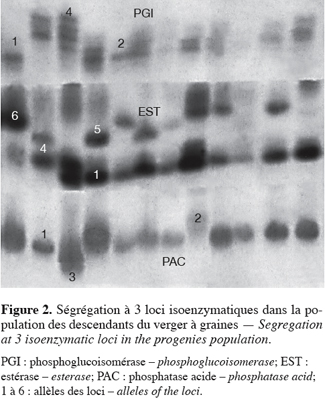
19Les loci PGI (E.C. : 5.1.3.9), LAP (E.C. : 3.4.11.1.) et EST (E.C. : 3.1.1) comportent chacun 4 allèles alors que ADH (E.C. : 1.1.1.1.) et PAC (E.C. : 3.1.3.2.) ont respectivement 2 et 3 allèles. Dans cette population parentale, seul l’allèle 3 du locus PAC (E.C. : 3.1.3.2.) (Figure 2) est rare puisque sa fréquence allélique est inférieure à 5 % (Tableau 3). L’allèle 3 de ce locus n’avait jamais été répertorié avant cette étude. Cet allèle a été mis en évidence sur les arbres du génotype codé 142.
20L’hétérozygotie moyenne de la population totale du verger à graines qui est de 0,35 pour l’ensemble des 5 loci considérés, apparaît inférieure à l’hétérozygotie théorique qui a été estimée à 0,55. De ce fait, l’index de fixation de cette population évalué à 0,36 (Tableau 3) indique un déficit en hétérozytes de 36 % par rapport à l’équilibre de Hardy-Weinberg. La fréquence d’hétérozygotes augmente des échantillons d’arbres-mères aux descendants. Cette fréquence passe de 0,33 à 0,44 en 1998 et de 0,34 à 0,44 en 1999 (Tableau 3) et le taux d’hétérozygotes des descendants est également supérieur à celui de toute la population du verger à graines. L’étude de l’index de fixation, montre que les échantillons d’arbre-mères présentent un déficit de 20 et de 29 % en hétérozygotes respectivement en 1998 et en 1999.
21Dans la population de descendants, le nombre total d’allèles est respectivement de 17 et 16 en 1998 et en 1999 (Tableau 4) et l’hétérozygotie observée est sensiblement égale à l’hétérozygotie théorique sur les deux années. Ainsi, dans cette population, l’index de fixation est faible et équivaut à 0,02 et à 0,04 respectivement en 1998 et 1999. Ces résultats montrent que le déficit en hétérozygotes de la population des descendants par rapport à l’équilibre de Hardy Weinberg est de 2 % en 1998 et de 4 % en 1999. L’index de fixation diminue donc fortement de la population de géniteurs du verger à graines à celle de leurs descendants issus de pollinisation libre. L’index de fixation des descendants n’est pas significativement différent de zéro. La consanguinité dans la population de descendants peut être alors négligée et les croisements se font de façon panmictique entre les arbres du verger à graines.
3.3. Paramètres du mode de reproduction
22Les paramètres du mode de reproduction estimés dans le verger à graines montrent que la proportion de descendants issus de croisements entre arbres de différents génotypes ou taux d’allofécondation est très importante. Elle est égale à 0,97 et 0,96 respectivement en 1998 et en 1999. Le taux d’allofécondation est ainsi peu variable entre les deux années mais reste cependant différent de l’allofécondation stricte c’est-à-dire 1 (Tableau 5). Le taux d’autofécondation qui s’établit à 0,3 en 1998 et à 0,4 en 1999 n’est donc pas négligeable. La valeur de la consanguinité biparentale estimée à 0,02 en 1998 et à 0,04 en 1999 suggère l’existence d’un faible appariement génétique entre arbres-mères du verger. La corrélation de paternité évaluée à 0,17 et 0,27 respectivement en 1998 et en 1999 est statistiquement différente de 0 et de 1. Ces valeurs indiquent que l’hétérozygotie élevée des descendants est due à la participation de plusieurs géniteurs mâles à la pollinisation des arbres du verger à graines. La corrélation d’allofécondation, respectivement égale à 0,4 en 1998 et à 0,45 en 1999, montre que le taux d’allofécondation est variable entre les familles des génotypes du verger à graines. Dans ces familles, le niveau de l’allofécondation se situe entre 0,83 et 1 sur l’ensemble des deux années (Tableau 6).
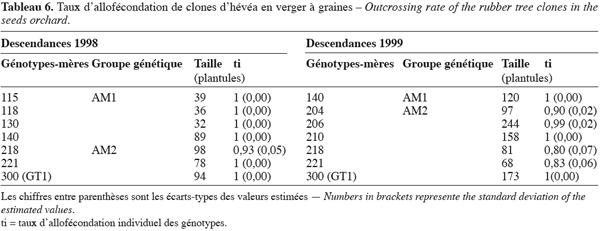
23Parmi les génotypes analysés, les géniteurs du groupe AM1 se caractérisent par une allofécondation stricte ce qui n’est pas le cas des génotypes de AM2 dont certains présentent un taux non nul d’autofécondation. Ce sont les génotypes codés 218 en 1998 et 204, 206, 218 et 221 en 1999 (Tableau 6). Sur les génotypes appartenant aux deux échantillons d’arbres-mères choisis, une diminution du taux d’allofécondation de 1998 à 1999 dans les familles des génotypes 218 et 221 a été observée. Ce taux est passé de 0,93 % à 0,81 pour le génotype 218 et de 1 à 0,83 pour le génotype 221, soit une réduction respective de 0,12 et de 0,17. Dans la famille du génotype 140, le taux d’allofécondation est stable quelle que soit l’année. Par ailleurs, aucune graine du génotype GT1 codé 300 n’est issue d’autofécondation, ce qui corrobore l’étude de Leconte (1984) qui a montré la stérilité mâle de ce génotype.
4. Discussion
24L’étude de la conformité clonale réalisée dans la population parentale sur les individus de même génotype a révélé des erreurs de greffage dans la mise en place du verger à graines. Certains arbres n’étaient pas conformes à leur présumé génotype. S’ils venaient à participer à la pollinisation, cela s’apparenterait à une contamination du verger à graines par du pollen extérieur. En effet, la mise en place d’un verger à graines répond à un objectif d’amélioration des populations ou de production de graines de bonne qualité. Le choix du matériel est alors fonction de l’objectif visé. Ainsi, la contamination d’un verger à graines par du pollen extérieur provoquerait une réduction des gains génétiques espérés (Greenwood, Rucker, 1985 ; Caron, Leblanc, 1992).
25L’hétérozygotie estimée à 0,35 dans la population parentale est importante puisqu’elle est supérieure à la moyenne de l’hétérozygotie des espèces forestières tropicales qui est de 0,22 (Hamrick, Godt, 1989). Cependant, la consanguinité apparaît élevée parmi les arbres du verger à graines. Ce taux élevé de la consanguinité dans la population parentale du verger à graines pourrait résulter des croisements consanguins de l’hévéa en forêt amazonienne. En effet, De Paiva (1994) ont estimé à 0,36 le taux d’autofécondation de l’hévéa en Amazonie et l’ont attribué à une structuration familiale des peuplements étudiés. De plus, la structuration de cette population sauvage d’hévéa en deux groupes génétiques suppose qu’ils ont évolué différemment dans leur habitat naturel. De sorte que le taux élevé de consanguinité noté dans cette population pourrait également s’expliquer par un effet Walhund. Dans le verger à graines, l’augmentation de l’hétérozygotie dans les descendants issus de pollinisation libre, suggère des croisements entre des arbres de génotypes différents. La consanguinité notée dans la population parentale s’annule au bout d’une génération car les croisements seraient panmictiques. Selon Khasa et al. (1993), la réduction de la consanguinité des parents aux descendants permet d’améliorer de façon significative les gains génétiques lors des tests de descendants en verger à graines. De même Baradat (1982) a également souligné l’importance des croisements panmictiques qui réduisent la consanguinité des parents aux descendants dans l’évaluation des géniteurs en pollinisation libre. Ainsi, les tests de descendants à réaliser dans ce verger à graines permettraient une meilleure évaluation des géniteurs en présence. Cependant, si le marqueur génétique et la méthodologie utilisés permettent bien de caractériser les croisements au niveau de la population, ils ne peuvent donner accès au nombre moyen de pollinisateurs d’un arbre. Pourtant, cette donnée apparaît capitale dans l’estimation de la valeur génétique des arbres pollinisés. En effet, plus le nombre de pollinisateurs d’un arbre est élevé et les contributions paternelles équilibrées, plus juste sera sa valeur génétique estimée.
26Pendant les deux campagnes de pollinisation libre dans le verger à graines, le taux d’allofécondation a été en moyenne estimé à 96 %. Le taux très élevé de l’allofécondation, confirme le caractère allogame de l’hévéa comme l’a souligné Compagnon (1986). En verger à graines, le taux de l’allofécondation apparaît plus important que celui estimé par Sunderasan et al. (1994) et par Yeang, Chevallier (1999) en plantation, sur des parcelles monoclonales d’hévéa. Dans cette situation, l’allofécondation peut-être assimilée globalement au flux de gènes entre des parcelles monoclonales voisines.
27En forêt amazonienne, centre d’origine de l’hévéa, De Paiva (1994) a estimé le taux d’allofécondation de l’hévéa à 64 % et a attribué l’importance de l’autofécondation qui est de 36 % principalement à la carence en allopollen sur les ovules à cause de la faible densité et la structuration familiale des peuplements naturels. L’auto-incompatibilité caractérisant la majeure partie des espèces forestières est donc levée chez l’hévéa en absence d’allopollen. Le mécanisme de cette auto-incompatibilité serait alors similaire à celle du cacaoyer (Lanaud et al., 1987).
28Le taux élevé de l’allofécondation observé en verger à graines serait lié d’une part à la forte densité de plantation et à la diversité génétique des arbres et d’autre part à la disponibilité de l’allopollen. De sorte qu’au cours de la pollinisation, les ovules des arbres reçoivent une importante quantité d’allopollen qui les féconde préférentiellement. Le dispositif de plantation, qui n’a pas favorisé la proximité entre arbres de même génotype aurait également contribué au taux élevé d’allofécondation observé.
29Les résultats de cette étude ont en outre montré que le taux d’allofécondation était dans l’ensemble constant sur les deux années malgré la différence des conditions climatiques. En effet, pendant la période de pollinisation, il est tombé 74 mm et 182 mm de pluie respectivement en 1998 et en 1999 (Lidah, 2005). Ainsi, le taux d’allofécondation dans le verger à graines apparaît peu sensible à la variation des conditions climatiques. Or, El Kassaby et al. (1988) et Madéore (1995) ont suggéré que l’allofécondation est un caractère quantitatif dont l’expression est modulée par les facteurs environnementaux. La faible variation du taux d’allofécondation du verger à graines pourrait encore s’expliquer par la disponibilité de l’allopollen d’une part et par l’importance sur le site de l’entomofaune intervenant dans la pollinisation de l’hévéa d’autre part.
5. Conclusion
30L’étude de la pollinisation libre de l’hévéa permettra de définir à terme, un schéma de sélection récurrente de l’hévéa en verger à graines. Les principaux résultats auxquels elle a abouti, à savoir l’importance du taux d’allofécondation, la diversité élevée dans la population des descendants et la réduction de l’index de fixation de la population parentale aux descendants sont encourageants dans la perspective de l’amélioration des populations sauvages d’hévéa. L’importance de l’allofécondation et de la diversité augure d’un bon brassage entre les géniteurs du verger à graines. La réduction de la consanguinité des parents aux descendants assurera une meilleure évaluation des géniteurs en présence sur test de descendance. Toutefois en pollinisation libre, les marqueurs isozymes ne nous permettent pas d’avoir accès au nombre moyen de pollinisateurs d’un arbre. De ce fait, cette étude pourrait être complétée par l’utilisation de marqueurs moléculaires tels les microsatellites en analyse de la paternité. Ce qui permettrait de déterminer des plans de croisements pour une meilleure évaluation des géniteurs utilisés.
31Remerciements
32Sincères remerciements au CNRA (Centre National de Recherche Agronomique), Station de Bimbresso de Côte d’Ivoire et au programme Hévéa du CIRAD (Centre de Coopération Internationale en Recherche Agronomique pour le Developpement) à Montpellier.
Bibliographie
Baradat P. (1982). Génétique quantitative. Modèles statistiques et génétiques de base. Fasicule I. Bordeaux, France : INRA, 203 p.
Baudoin L., Baril C., Clément-Demange A., Leroy T., Paulin D. (1997). Recurrent selection of tropical tree crops. Euphytica 96, p. 101–114.
Caron GE., Leblanc R. (1992). Pollen contamination in a small black spruce seedlineg seed orchard during three consecutive years. For. Ecol. Manag. 53, p. 245–261.
Compagnon P. (1986). Le caoutchouc naturel : biologie, culture, production. Collection « Techniques agricoles et productions tropicales ». Paris : Maisonneuve et Larose, 595 p.
Clément-Demange A., Nicolas D., Légnaté H., Rivano F., Le Guen V., Gnagne M., Chapuset T. (1995). Hévéa, stratégies de sélection. Rech. Dév. 2 (3), p. 5–18.
Chevalier MH. (1988). Genetic variability of Hevea brasiliensis germplasm using isozyme markers. J. Nat.Rub. Res. 3 (1), p. 42–53.
Chevallier MH., Lebrun P. (1988). Starch and poly-acrylamide gel electrophoresis of Hevea brasiliensis: a laboratory manual. Montpellier, France : IRCA-CIRAD Publishers, 33 p.
Dagnelie P. (1998). Statistique théorique et appliquée. Vol. 2. Inférence statistique à une et deux dimensions. Bruxelles : De Boeck Université, 651 p.
Demarly Y. (1977). Génétique et amélioration des plantes. Collection Sciences Agronomiques. Paris, France : Masson, 287 p.
De Paiva JRD. (1994). Genetics of rubber tree (Hevea brasiliensis). Mating system. Silvae Genet. 43, p. 373–376.
El-Kassaby YA., Ritland K., Fashler AMK., Devitt WJP. (1988). The role of reproductive phenology upon the mating system of a Douglas-fir seed orchard. Silvae Genet. 37, p. 76–82.
Greenwood MS., Rucker R. (1985). Estimating pollen contamination in loblolly pine orchards by pollen trapping. Compte rendu de la 18e conférence sur l’amélioration génétique des arbres du Sud, 21-23 mai. Long Beach, Miss., p. 179–186.
Hamrick JL., Godt MJW. (1989). Allozyme diversity in plant species. In Brown AHD., Clegg MT., Kahler AL., Weir BS. (eds). Plant population genetics, breeding and genetic resources. Sunderland, MA, USA: Sinauer Associates, p. 43–64.
Khasa PD., Cheliak WM., Bousquet J. (1993). Mating system of Racosperna auriculiform in a seed production area in Zaïre. Can. J. Bot. 71, p. 779–785.
Lanaud C., Sounigo O., Amefia YK., Paulin D., Lachenaud PH., Clément D. (1987). Nouvelles données sur le fonctionnement du système d’incompatibilité du cacaoyer. Café Cacao Thé 31 (4), p. 267–281.
Leconte A. (1984). La reproduction sexuée de l’Hevea brasiliensis : une approche histologique et expérimen-tale. Doctorat 3e cycle. Univ. Sc. Languedoc, Montpellier, France, 68 p.
Légnaté H., Clément-Démange A. (1991). Pollinisation artificielle et amélioration génétique de l’hévéa en Côte d’Ivoire. Caoutchouc Plast. 136/91, p. 99–103.
Lidah YJ. (2005). étude de la fécondation libre d’une population sauvage d’hévéa (Hevea brasiliensis Muell. Arg.) en verger à graines. Thèse de Doctorat 3e cycle. Abidjan, Côte d’Ivoire : Université de Cocody, UFR Biosciences, 129 p.
Madéore C. (1995). Régime de reproduction dans un verger à graines de teck (Tectona grandis L.f.) en Côte d’Ivoire. DEA. école Normale Supérieure d’Horticulture de Versailles. Montpellier, France : CIRAD-Forêt, 54 p.
Nei M. (1987). Molecular evolutionary genetics. New York: Columbia University Press.
Nicolas D. (1979). Quelques données sur la fécondation et la pollinisation artificielle chez Hevea brasiliensis. Rapport de recherche 28/79. Montpellier, France : IRCA, 35 p.
Nicolas D. (1981). Prospection et récolte de matériel végétal d’hévéa dans la forêt amzonienne. Le cas de l’état de l’Acre au Brésil. Rapport de mission. Kuala Lumpur, Malaisie : IRRDB, 38 p.
Rao BS. (1961). Pollination of Hevea in Malaya. J. Rubb. Res. Inst. Malaya 17, p. 14–18.
Ritland K., Jain S. (1981). A model for the estimation of outcrossing rate and gene frequencies using n dependent loci. Heredity 47, p. 35–52.
Ritland K., El-Kassaby (1985). The nature of inbreeding in a seed orchard of a Douglas-fir sa show by an efficience multilocus model. Theor. Appl. Genet. 71, p. 375–384.
Ritland K. (1994). Computer program for estimating plant mating system. J. Heredity 81, p. 235–237.
Sunderasan E., Wickneswari R., Azziz MZ., Yeang HY. (1994). Incidence of self and cross-pollination in two Hevea brasiliensis clones. J. Nat. Rub. Res. 9 (4), p. 253–257.
Wright S. (1964). The interpretation of population structure by F-statistics with the special regard to the systems of mating. Evolution 19, p. 395–420.
Yeang YH., Chevallier MH. (1999). Range of Hevea brasiliensis Pollen dispersal estimate by esterase isozyme markers. Ann. Bot. Com. 84, p. 681–684.
Pour citer cet article
A propos de : Yehili Justin Lidah
Laboratoire de Génétique. Université de Cocody Abidjan 22. BP 582 Abidjan 22 (Côte d’Ivoire). E-mail : justinlidah@yahoo.fr
A propos de : Assanvo Simon-Pierre N’Guetta
Laboratoire de Génétique. Université de Cocody Abidjan 22. BP 582 Abidjan 22 (Côte d’Ivoire).
A propos de : Mondeil Fanjavola
Laboratoire de Génétique. Université de Cocody Abidjan 22. BP 582 Abidjan 22 (Côte d’Ivoire).
A propos de : André Clément-Demange
CIRAD Cultures Pérennes. Programme Hévéa. Avenue Agropolis. F-34398 Montpellier. Cedex 5 (France).
A propos de : Goud Margueritte Rodier
CIRAD Cultures Pérennes. Programme Cacao AGETROP. Avenue Agropolis. F-34398 Montpellier. Cedex 5 (France).