Optimalisation de l'extraction d'ADN génomique de la morelle jaune (Solanum elaeagnifolium Cav.), une plante invasive des milieux cultivés en région méditerranéenne
European Biological Control Laboratory-ARS-USDA. Campus International de Baillarguet. CS90013. Montferrier sur Lez. F-34980 Saint Gély du Fesc (France).
European Biological Control Laboratory-ARS-USDA. Campus International de Baillarguet. CS90013. Montferrier sur Lez. F-34980 Saint Gély du Fesc (France). E-mail : mcbon@ars-ebcl.org
European Biological Control Laboratory-ARS-USDA. Campus International de Baillarguet. CS90013. Montferrier sur Lez. F-34980 Saint Gély du Fesc (France).
Reçu le 26 mai 2010, accepté le 30 aout 2010
Résumé
L'origine géographique d'une plante invasive des milieux cultivés en région méditerranéenne, la morelle jaune, Solanum elaeagnifolium Cav. (Solanaceae), doit être identifiée par l'analyse des ressemblances génétiques au moyen de marqueurs microsatellites entre populations natives et introduites. Au préalable, une méthode efficace et peu couteuse d'extraction d'ADN génomique à partir de feuilles déshydratées et riches en polysaccharides a été recherchée. Le protocole originel de Doyle utilisant le détergent cationique, CTAB, a été optimisé avec succès pour permettre l'extraction d'ADN de qualité significativement supérieure (rapport A260/280 de 1,8 et A260/230 de 1,7 à 2). Les rendements en quantité d'ADN extrait avec ce protocole ont été supérieurs à ceux obtenus au moyen d'un kit commercial pour une qualité comparable de l'ADN extrait. L'optimalisation du protocole a été validée par l'amplification de plusieurs loci microsatellites originellement développés sur l'aubergine, Solanum melongena L.
Abstract
Optimization of the genomic DNA extraction method of silverleaf nightshade (Solanum elaeagnifolium Cav.), an invasive plant in the cultivated areas within the Mediterranean region. The geographical origin of an invasive plant in the cultivated area within the Mediterranean region, silverleaf nightshade, Solanum elaeagnifolium Cav. (Solanaceae) should be identified through the analysis of genetic similarities between native and introduced populations using microsatellite markers. Beforehand, an effective and less costly method for extracting genomic DNA from dehydrated and polysaccharide-rich leaves was investigated. The original Doyle's protocol based on the use of a cationic detergent, CTAB, was successfully optimized in order to extract high quality DNA (A260/280 ratio of 1.8; A260/230 ratio of 1.7 to 2). The DNA yields obtained with this new protocol were higher than those obtained with a commercial kit, although quality of the DNA extracted was comparable. The improvement of the protocol was further proven by the amplification of several microsatellite loci first developed in eggplant, Solanum melongena L.
1. Introduction
1L'intensification des échanges commerciaux, notamment par transports aériens et maritimes, ainsi que les migrations humaines contribuent à la dissémination accidentelle, ou non, d'espèces hors de leur aire d'origine (Monty et al., 2009). Les populations d'espèces, dites allochtones car introduites dans des nouveaux territoires, peuvent parfois devenir, en l'absence de leur cortège parasitaire d'origine, des envahisseurs préjudiciables aux écosystèmes et à la biodiversité (Bossdorf et al., 2005; Sforza et al., 2007). De fait, les milieux cultivés et anthropisés constituent les premiers habitats colonisés par les plantes allochtones. L'étude européenne DAISIE (Delivering Alien Invasive Species in Europe) achevée en 2008 montre que 58 % des plantes naturalisées en Europe sont présentes dans les milieux cultivés (Chauvel et al., 2009). Le cas de Solanum elaeagnifolium Cav. (Solanaceae), native des États-Unis (sud-ouest des États-Unis, nord du Mexique et potentiellement Argentine/Chili) illustre ce constat. Sa rapide dispersion au cours du 19e siècle en Europe (14 pays) dont le bassin méditerranéen, et ses impacts négatifs tant sur l'économie (compétition par rapport aux plantes cultivées, plante-hôte de certains virus des solanacées cultivées), sur l'environnement (réduction de la biodiversité) et la santé humaine et animale ont conduit l'Organisation Européenne et Méditerranéenne pour la Protection des Plantes (OEPP) à la classer comme organisme de quarantaine (Buck et al., 1960 ; Boyd et al., 1984 ; Ehret, 2006 ; Mekki, 2007 ; OEPP, 2007 ; Tscheulin et al., 2009). Un programme de lutte biologique classique est envisagé pour certains pays de la région méditerranéenne dont les pays du Maghreb et la Grèce (Bouhache et al., 2004 ; Sforza et al., 2007). La lutte biologique classique est définie comme l'introduction intentionnelle d'un agent de lutte biologique exotique, ayant coévolué avec la plante, pour un établissement permanent et un contrôle à long terme (Eilenberg et al., 2001). Pour savoir si les populations invasives ont subi des changements évolutifs liés à la colonisation et pour trouver les ennemis naturels phytophages les plus spécifiques et efficaces, il est nécessaire de connaitre l'origine exacte des populations invasives et d'évaluer l'étendue de la diversité génétique des populations à contrôler (Bossdorf et al., 2005 ; Ward et al., 2009). Les analyses de la diversité génétique, de la structuration des populations ainsi que de l'évaluation des échanges entre les populations (migration, flux géniques) sont classiquement appréhendées par des marqueurs moléculaires neutres comme les microsatellites (Roderick et al., 2003 ; Ward et al., 2009). Les microsatellites, séquences répétées en tandem de période de une à six paires de bases, répartis sur l'ensemble du génome sont hautement polymorphes et codominants (Li et al., 2002). Leur utilisation en génotypage via une PCR (Polymerase Chain Reaction) nécessite entre autre une bonne qualité d'ADN génomique. Seules, des études en phylogénie moléculaire font état d'extraction d'ADN de morelle jaune à partir de feuilles fraiches ou déshydratées en suivant le protocole de Doyle et al. (1987) sans indication de la qualité obtenue (Bohs et al., 2001 ; Weese et al., 2007). Des essais préliminaires ont montré qu'il était nécessaire d'optimiser la technique d'extraction d'ADN compte tenu des facteurs suivants :
2– la quantité et la qualité de l'ADN extrait en vue du génotypage,
3– le cout vu le nombre important d'échantillons à extraire,
4– un protocole utilisable par différents laboratoires dont certains sont peu équipés,
5– la nécessité de travailler sur du matériel déshydraté.
6Dans cette étude, nous avons développé un protocole simple, efficace permettant d'extraire de façon homogène de l'ADN génomique de qualité à partir de feuilles déshydratées de morelle jaune.
2. Matériel et méthodes
2.1. Matériel végétal
7L'extraction de l'ADN génomique de la morelle jaune a été réalisée à partir de plantes récoltées sur le terrain, dans l'aire d'introduction, soit six accessions grecques (Thessalonique, Lesvos A et B, Leukada et Péloponnèse A et B) et dans l'aire native, soit trois accessions texanes, USA (LaVilla, Faysville et Bayside). Seules, les dernières paires de feuilles sont prélevées puis déshydratées par Silica gel sans cobalt pendant au moins une semaine, avant d'être rapportées au laboratoire pour y être conservées avec le Silica gel en quarantaine.
2.2. Méthodes
8Extraction de l'ADN. Le protocole d'extraction de référence est celui d'un kit commercial, Qiagen DNeasy Plant Mini Kit (Courtaboeuf, France), sans modification. Il limite la quantité de matériel sec à extraire à 20 mg. Le protocole de base non commercial est celui de Doyle et al. (1987) à base de détergent cationique CTAB (bromure d'hexadécyltriméthyl ammonium) déjà utilisé par Murray et al. (1980). Les paramètres testés pour optimiser ce protocole sont la concentration en chlorure de sodium et en PVP du tampon d'extraction, le mode de précipitation et de resuspension de l'acide nucléique. Le protocole d'extraction optimisé est décrit comme suit, le tampon d'extraction étant composé de 2 % (p/v) de CTAB ; 1,4 M NaCl ; 20 mM Na2EDTA ; 100 mM Tris-HCl (pH 8) et 1 % de bisulfite de sodium ajouté extemporanément :
9– environ 20 mg de feuilles déshydratées mises en suspension dans 1 ml de tampon d'extraction préchauffé à 60 °C sont broyées dans un mortier autoclavé,
10– le broyat est incubé à 60 °C au bain-marie pendant 1 h avec une agitation manuelle et fréquente par inversion des tubes,
11– après une centrifugation rapide, 500 µl d'un mélange phénol/chloroforme/alcool isoamylique 25:24:1 sont ajoutés au surnageant, l'ensemble est agité, centrifugé à 10 000 g pendant 10 min à 4 °C,
12– à la phase aqueuse sont ajoutés 500 µl d'un mélange chloroforme/alcool isoamylique 24:1, l'ensemble après agitation est centrifugé à 10 000 g pendant 10 min à 4 °C,
13– à la phase aqueuse, sont ajoutés 2/3 de volume d'isopropanol, le mélange est conservé à -20 °C pendant 30 min;
14– après précipitation, l'ensemble est centrifugé à 10 000 g pendant 10 min à 4 °C,
15– après élimination du surnageant, le culot est séché dans un évaporateur concentrateur centrifuge sous vide (Speed Vac Savant Instruments, Farmingdale, USA) pendant 5 min et resuspendu en présence de 500 µl de NaCl 1M et de RNase A à la concentration finale de 800 µg.ml-1 pendant 30 min à 37 °C;
16– 500 µl d'un mélange chloroforme/alcool isoamylique 24:1 sont ensuite ajoutés, les tubes agités manuellement et centrifugés à 10 000 g pendant 10 min à 4 °C,
17– à la phase aqueuse sont ajoutés 2/3 de volume d'isopropanol, le mélange est conservé à -20 °C pendant 30 min,
18– après précipitation, l'ensemble est centrifugé à 10 000 g pendant 10 min à 4 °C,
19– après élimination du surnageant, le culot d'ADN est séché à l'évaporateur concentrateur centrifuge sous vide pendant 5 min et resuspendu dans 30 µl de TE (10m M Tris (pH 8), 20 mM Na2EDTA) toute la nuit à 4 °C. Un témoin d'extraction sans matériel biologique est systématiquement inclus dans la procédure.
20La quantité et la qualité de l'ADN (1,5 µl) sont estimées par dosage au spectrophotomètre Nanodrop (Thermo Scientific NanoDropTM8000).
21Analyses statistiques. Pour chacun des protocoles et accessions (grecques ou texanes) testés, les quantités en ADN, les ratios de densités optiques A260/A280 et A260/A230 sont comparés par analyse statistique à l'aide du logiciel XLstat (Addinsoft). La normalité des données est testée avec un test de Shapiro. Pour les variables ne suivant pas une loi normale (p > 0,01), les données sont traitées à l'aide du test non paramétrique de Kruskal-Wallis avec correction de Bonferroni qui permet de garantir un risque alpha d'erreur de 5 % lors de comparaisons multiples.
22Amplification des marqueurs microsatellites. La qualité de l'ADN est estimée par l'amplification de 16 loci microsatellites dont EM131 développés par Nunome et al. (2003) sur l'aubergine, Solanum melongena L., espèce phylogénétiquement très proche de la morelle jaune (Weese et al., 2007). Le volume pour la réaction PCR finale est de 10 µl et contient du Mutiplex Master Mix Qiagen 1X, 0,3 µM de l'amorce sens et antisens et 10 ng d'ADN. Les conditions de la PCR réalisée dans un thermocycleur Perkin Elmer 9700 sont les suivantes : dénaturation à 95 °C et activation de la Taq pendant 5 min et 30 cycles de 30 sec à 94 °C, 30 sec à 52 °C et 1 min à 72 °C, suivi d'une élongation finale à 72 °C pendant 7 min. Le produit PCR est déposé sur gel d'agarose à 2 % en présence de SybrSafe. Les gels sont visualisés sous UV à 254 nm au transilluminateur (Fisher Bioblock Scientific).
3. Résultats et discussion
23De nombreux protocoles d'extraction d'ADN de plantes existent dans la littérature (Varma et al., 2007) dont les plus cités historiquement sont ceux de Murray et al. (1980), Dellaporta et al. (1983), Doyle et al. (1987) et Rogers et al. (1988). Même si ces méthodes ont prouvé leur efficacité, elles sont rarement universellement applicables en tant que telles à tous les taxons et types de matériels. Dans cette étude, nous avons cherché à optimiser certaines étapes du protocole de Doyle et al. (1987) en vue de proposer une méthode d'extraction d'ADN de qualité, relativement simple, peu couteuse, applicable par plusieurs coopérateurs de pays où la morelle jaune est un problème pour l'écologie et l'agriculture.
24Cette mise au point a porté sur 180 extractions à partir d'accessions différentes (Texas - États-Unis, Grèce). Les quantités d'ADN ainsi que les ratios d'absorbance 260/280 et 260/230 sont comparés entre le kit commercial, le protocole de référence de Doyle et al. (1987) et le protocole optimisé (Tableau 1).
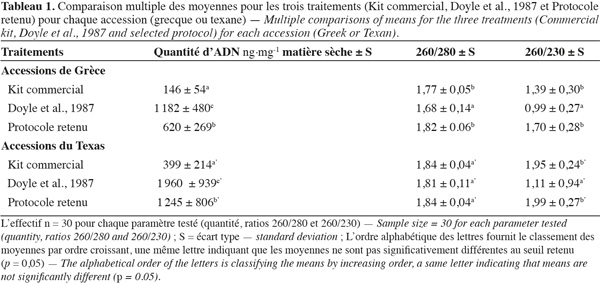
25Les ajustements apportés au cours de cette étude ont permis l'obtention d'ADN en quantités significativement plus importantes avec le nouveau protocole ou celui de Doyle par comparaison au kit commercial et ce, quelle que soit l'accession. Les ADN extraits selon Doyle présentent d'importantes quantités d'ARN en dépit du traitement RNase.
26Le ratio 260/280 est de l'ordre de 1,8 avec le protocole proposé et ce, quel que soit le type d'accession. Il correspond à une valeur attendue de l'ADN purifié de 1,8 à 2 (Sambrook et al., 1989). Seules les valeurs des populations grecques obtenues pour le traitement Doyle sont significativement inférieures, indiquant une possible contamination protéique.
27Le ratio 260/230 varie de 1,4 à 1,95 pour le kit commercial et de 0,99 à 1,11 pour celui de Doyle, indiquant une forte contamination par des composés comme les polysaccharides qui absorbent à 230 nm (Varma et al., 2007). Quelle que soit l'accession considérée, il est toujours supérieur et proche de la valeur attendue 2 à 2,2 pour le protocole optimisé en comparaison au kit commercial, bien que non significativement différent. Quelle que soit l'accession considérée, le traitement retenu a permis d'obtenir de façon plus reproductible des ratios 260/280 et 260/230 proches des valeurs théoriques attendues. Le protocole proposé est le plus approprié pour une application de génotypage d'un échantillonnage très divers.
28Cette qualité de l'ADN extrait a aussi été testée par amplification et visualisation sur gel de loci microsatellites dont EM131 (Figure 1).

29Les paramètres testés pour optimiser ce protocole par rapport à celui de Doyle et al. (1987) sont listés dans le tableau 2.
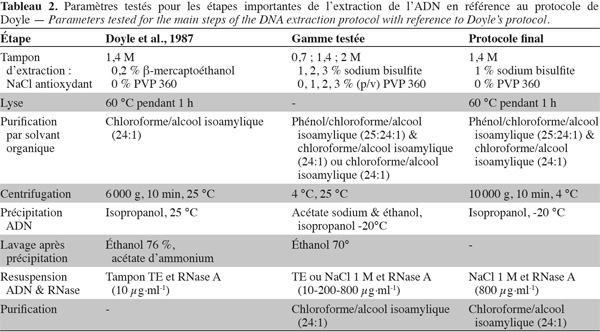
30Les extraits initiaux obtenus selon le protocole de Doyle et al. (1987) contenaient de grandes quantités d'ARNs, de protéines, de polysaccharides et dans une moindre mesure des pigments, autant de composants qui interfèrent avec l'ADN et restent difficiles à éliminer. La morelle jaune est riche en glycoalcaloïdes comme la solanine et solasonine (Boyd et al., 1984 ; Knight et al., 2003) mais aussi de polysaccharides comme d'autres Solanum dont S. nigrum L. (Stankiewicz et al., 2000). Des antioxydants sont communément utilisés pour limiter l'action des polyphénols qui, après oxydation, sont liés de façon covalente aux protéines et acides nucléiques au cours de l'homogénéisation. Chez la tomate, Peterson et al. (1997) avaient déjà relaté la présence de quantités importantes de polyphénols. L'un des antioxydants les plus utilisés, le β-mercaptoéthanol, a été avantageusement remplacé par le bisulfite de sodium, non toxique et moins couteux (Loomis, 1974 ; Aljanabi et al., 1999). L'ajout de PVP, aussi recommandé dans plusieurs protocoles pour l'adsorption des composés phénoliques à pH bas, n'est pas apparu nécessaire dans cette étude (Peterson et al., 1997). Pour des concentrations élevées, le PVP co-précipite avec l'ADN et apparait alors comme un contaminant (Varma et al., 2007). Les polysaccharides sont particulièrement problématiques car co-précipitent avec l'ADN. Ils rendent les pipetages et la migration sur gel difficiles, causent des anomalies dans les cinétiques de réassociation et interfèrent avec les activités enzymatiques telles que les ligases, nucléases (Varma et al., 2007). L'addition de NaCl à une concentration supérieure à 0,5 M avec le CTAB est connue pour éliminer les polysaccharides (Fang et al., 1992) et a une incidence sur les amplifications par PCR (Kawata et al., 2003). Les concentrations variant de 0,7 à 6 M dépendent de la plante (Puchoda et al., 2004). Les sels comme NaCl sont ajoutés pour moduler la concentration des cations dans le tampon d'extraction. Des extractions au phénol-chloroforme ont amélioré les quantités d'ADN extrait ainsi que leur qualité par rapport à un lavage chloroforme seul. Ces lavages avec des solvants organiques favorisent l'élimination des protéines dont la polarité dans ces phases organiques change. La centrifugation à 4 °C au lieu de 25 °C favorise l'obtention d'une interphase plus compacte entre les phases aqueuses et organiques, soit après le phénol ou le chloroforme, permettant une meilleure séparation. Ces résultats corroborent ceux obtenus sur le cotonnier par Benbouza et al. (2006). L'utilisation d'un mélange acétate d'ammonium/éthanol pour précipiter l'ADN entraine la formation d'un précipité blanchâtre, y compris pour le témoin, attribuable au CTAB, bien que celui-ci soit relativement soluble dans l'éthanol. Il a été avantageusement remplacé par l'isopropanol, réduisant le précipité blanc et a permis d'améliorer considérablement la qualité de l'ADN. Malgré l'utilisation d'isopropanol en première précipitation, un précipité mucilagineux est toujours obtenu, à l'exception du témoin. Ces mucilages, de nature exopolysaccharidique, sont composés de polymères de glycoprotéines. Très présents dans plusieurs groupes de plantes, dont les plantes succulentes, ils sont facilement détectables en phase de resuspension dans le TE ou après précipitation par l'isopropanol (Cota-Sanchez et al., 2006). La contamination de l'ADN par ces mucilages induirait des amplifications aléatoires, voire inhiberait les PCR (Fang et al., 1992 d'après Cota Sanchez et al., 2006 ; Ghosh et al., 2009). La suspension de l'ADN dans une solution de NaCl 1 M plutôt que dans du TE a permis d'éliminer ce culot gélatineux. L'utilisation de tampon très salin pour la resuspension de l'ADN précipité permettrait non seulement de réduire la viscosité du mucilage, mais aussi augmenterait la spécificité de la RNase comme rapporté dans Chen et al. (2004) et Ghosh et al. (2009).
31Les modifications apportées au protocole de base de Doyle et al. (1987) ont permis de proposer un protocole d'extraction d'ADN de bonne qualité et de qualité constante pour des échantillons très divers en vue de génotypage par microsatellites d'un grand nombre de populations qui sera réalisé ultérieurement par analyse des fragments sur séquenceur capillaire. De plus, ce protocole s'avère relativement simple et moins couteux qu'un kit commercial.
32Remerciements
33Les auteurs remercient Javid Kashefi (EBCL, Thessalonique, Grèce) et Randy Coleman (USDA-ARS, USA) pour les récoltes et l'envoi du matériel végétal, Mélanie Jeanneau et Fatiha Guermache (EBCL) pour leurs conseils techniques et Dominique Coutinot (EBCL) pour l'obtention du permis d'importation et de détention de ce matériel de quarantaine (10LR013) et enfin, le Centre de Biologie et de Gestion des Populations (CBGP) à Montpellier en France pour l'utilisation de son Nanodrop.
34Abréviations
35CTAB : Bromure d'hexadécyltriméthyl ammonium
36PVP : Polyvinylpyrrolidone 360
Bibliographie
Aljanabi S.M., Forget L. & Dookun A., 1999. An improved rapid protocol for the isolation of polysaccharide and polyphenol-free sugarcane DNA. Plant Mol. Biol. Rep., 17, 1-8.
Benbouza H., Baudoin J.-P. & Mergeai G., 2006. Amélioration de la méthode d'extraction d'ADN au CTAB appliquée aux feuilles de cotonnier. Biotechnol. Agron. Soc. Environ., 10, 73-76.
Bohs L. & Olmstead R.G., 2001. A reassessment of Normania and Triguera (Solanaceae). Plant Syst. Evol., 228, 33-48.
Bossdorf O. et al., 2005. Phenotypic and genetic differentiation between native and introduced plant populations. Oecologia, 144, 1-11.
Bouhache M., Baye Y., Ameur A. & Taleb A., 2004. Quelques éléments de la biologie de la morelle jaune. In : 12e Colloque international sur la biologie des mauvaises herbes, 31.08-02.09.2004, Dijon, France, 20-31.
Boyd J.W., Murray D.S. & Tyrl R.J., 1984. Silverleaf nightshade, Solanum elaeagnifolium, origin, distribution, and relation to man. Econ. Bot., 38(2), 210-217.
Buck W.B., Dollahite J.W. & Allen T.J., 1960. Solanum elaeagnifolium, silver-leafed nightshade poisoning in livestock. J. Am. Vet. Med. Assoc., 137, 348-351.
Chauvel B. & Fried G., 2009. Dans la jungle des milieux cultivés. Pour la Science, 65, 32-37.
Chen R.H. & Chen W.Y., 2004. Rheological properties of the water soluble mucilage of a green laver, Monostroma nitidum. J. Appl. Phycol., 13, 481-488.
Cota-Sanchez J.H., Remarchuk K. & Ubayasena K., 2006. Ready-to-use DNA extracted with a CTAB method adapted for herbarium specimens and mucilaginous plant tissue. Plant Mol. Biol. Rep., 24, 161-167.
DAISIE (European Invasive Alien Species Gateway), 2008. Solanum elaeagnifolium, http://www.europe-aliens.org/speciesFactsheet.do?speciesId=, (08.10.2010).
Dellaporta S.L., Wood J. & Hicks J.B., 1983. A plant DNA minipreparation: version II. Plant Mol. Biol. Rep., 1(4), 19-21.
Doyle J.J. & Doyle J.L., 1987. A rapid DNA isolation procedure for small quantities of fresh leaf tissue. Phytochem. Bull., 19, 11-15.
Ehret P., 2006. Plantes exotiques envahissantes en France métropolitaine. Quelles observations pour quelles interventions ? In : AFFP, 1ère Conférence sur l'entretien des espaces verts, jardins, gazons, forêts, zones aquatiques et autres zones non agricoles, 11-12.10.2006, Avignon, France, 1-7.
Eilenberg J., Hajek A. & Lomer C., 2001. Suggestions for unifying the terminology in biological control. BioControl, 46, 387-400.
Fang G., Hammer S. & Grumet R., 1992. A quick and inexpensive method for removing polysaccharides from plant genomic DNA. Biotechniques, 13, 52-57.
Ghosh R., Paul S., Ghosh S.K. & Roy A., 2009. An improved method of DNA isolation suitable for PCR-based detection of begomoviruses from jute and other mucilaginous plants. J. Virol. Methods, 159, 34-39.
Kawata M. et al., 2003. Analysis of extraction buffer components from plant tissue by polymerase chain reaction. Anal. Biochem., 318, 314-317.
Knight A.P. & Walter R.G., 2003. A guide to plant poisoning of animals in North America. Jackson, WY, USA: Teton NewMedia Publisher, http://www.ivis.org, (08.04.2010).
Li Y.C. et al., 2002. Microsatellites: genomic distribution, putative functions and mutational mechanisms: a review. Mol. Ecol., 11, 2453-2465.
Loomis W.D., 1974. Overcoming problems of phenolics and quinones in the isolation of plant enzymes and organelles. Methods Enzymol., 3l, 528-545.
Mekki M., 2007. Biology, distribution and impacts of silverleaf nightshade (Solanum elaeagnifolium Cav.). EPPO Bull., 37, 114-118.
Monty A. & Mahy G., 2009. Évolution des traits d'histoire de vie lors des invasions végétales. Biotechnol. Agron. Soc. Environ., 13, 449-458.
Murray M. & Thompson W.F., 1980. Rapid isolation of high molecular weight plant DNA. Nucl. Acids Res., 8, 4321-4325.
Nunome T., Suwabe K., Iketani H. & Hirai M., 2003. Identification and characterization of microsatellites in eggplant. Plant Breeding, 122, 256-262.
OEPP/EPPO, 2007. Solanum elaeagnifolium. Data sheets on quarantine pests. EPPO Bull., 37, 236-245, http://www.eppo.org, (08.04.2010).
Peterson D.G., Boehm K.S. & Stack S.M., 1997. Isolation of milligram quantities of nuclear DNA from tomato (Lycopersicon esculentum), a plant containing high levels of polyphenolic compounds. Plant Mol. Biol. Rep., 15, 148-153.
Puchoda D., Shahnoo S.-U. & Khoyratty S., 2004. Genomic DNA extraction from Victoria amazonica. Plant Mol. Biol. Rep., 22, 195a-195j.
Roderick G. & Navajas M., 2003. Genes in new environments: genetics and evolution in biological control. Nat. Rev. Genet., 4, 889-899.
Rogers S.O. & Bendich A.J., 1988. Extraction of DNA from plant tissues. In: Gelvin S.B. & Schilperoort R.A., eds. Plant molecular biology manual, A6. Boston, MA, USA: Kluwer Academic Publishers, 1-10.
Sambrook J., Fritsch E.F. & Maniatis T., 1989. Molecular cloning. A laboratory manual. Vol.3. Cold Spring Harbor, NY, USA: Cold Spring Harbor Laboratory Press.
Sforza R. & Jones W.A., 2007. Potential for classical biocontrol of silverleaf nightshade in the Mediterranean bassin. EPPO Bull., 37, 156-162.
Stankiewicz M., Gadamski G. & Gawronski S.W., 2000. Genetic variation and phylogenetic relationships of triazine-resistant and triazine susceptible biotypes of Solanum nigrum – analysis using RAPD markers. Weed Res., 41, 287-300.
Tscheulin T., Petanidou T., Potts S.G. & Settele J., 2009. The impact of Solanum elaeagnifolium, an invasive plant in the Mediterranean, on the flower visitation and seed set of the native co-flowering species Glaucium flavum. Plant Ecol., 205, 77-85.
Varma A., Padh H. & Shrivastava N., 2007. Plant genomic DNA isolation: an art or a science. Biotechnol. J., 2, 386-392.
Ward S.M. & Jasienuck M., 2009. Review: sampling weedy and invasive plant populations for genetic diversity analysis. Weed Sci., 57, 593-602.
Weese T.L. & Bohs L., 2007. A three-gene phylogeny of the genus Solanum (Solanaceae). Syst. Bot., 32(2), 445-463.

